Simulér et ribosom — Virtual Ribosome
Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion


Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion
Virtual Ribosome bruges til at translatere DNA-sekvenser til proteinsekvenser. Ud over den direkte translation bruges Virtual Ribosome også til at analysere de seks mulige læserammer, da programmet kan finde den mest sandsynlige læseramme for DNA-sekvensen. Hvis man har en ukendt DNA-sekvens, er analysen af læserammer en stor fordel, da man ved at undersøge dem for den åbne læseramme kan finde protein-kodende gener i sekvenserne.
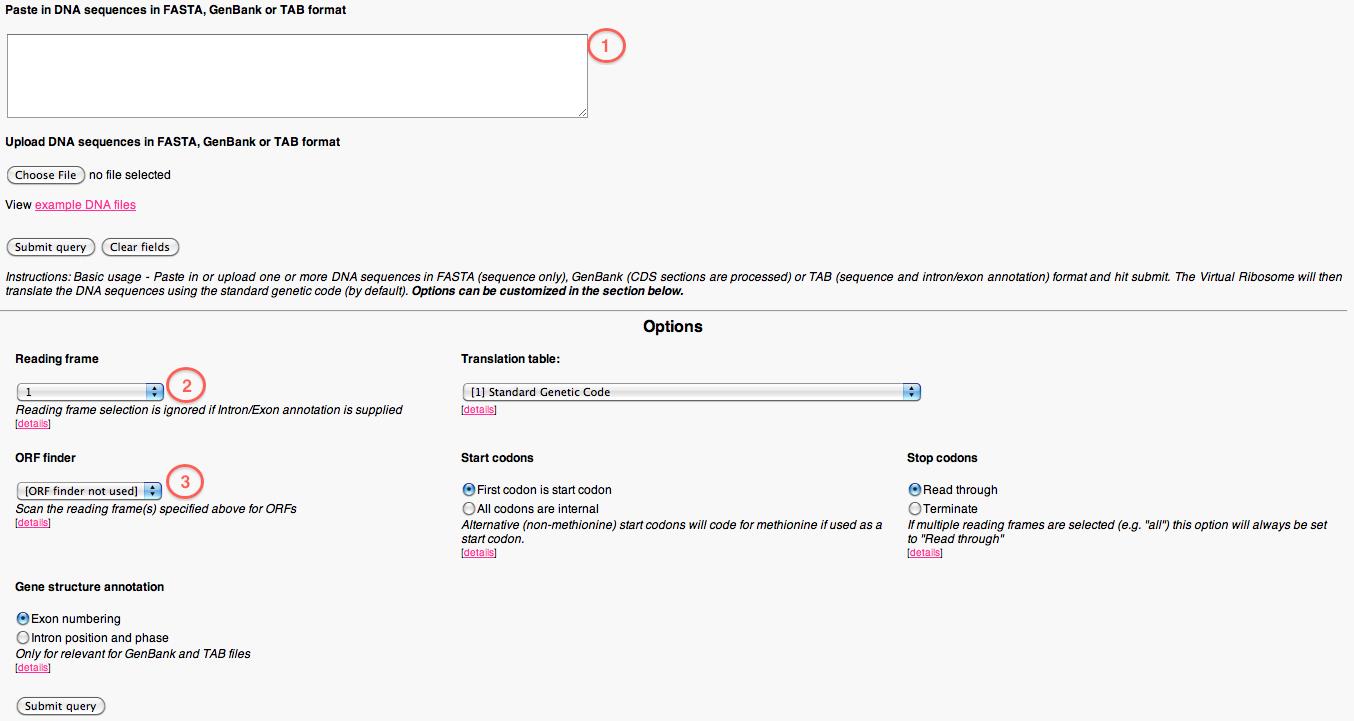
Figur 36. Startsiden for Virtual Ribosome.
Når man går til internetsiden for Virtual Ribosome, kommer man direkte ind på den startside, som kan ses i figur 36. Den DNA-sekvens, man ønsker at translatere, indtastes i felt 1 på figur 36, og den kan indtastes både med og uden “header” (se nedenfor).
En DNA-sekvens med header ser ud som nedenfor. Det er vigtigt at huske at skifte til en ny linje mellem headeren og selve nukleotid-sekvensen, da linjen adskiller dem:
>protein 1 (header)
AATGTGGACCTCAGTTGACTAGCAGTTAGGAGCACAGATGACAGTA
Et eksempel på en DNA-sekvens uden header:
ACCGGATGCGTAACAGATAGGGCACCCATGATGACGAACGTATAG
Sekvenser med headere af denne type kaldes for FASTA-filer.
Hvis der ikke ændres på parametrene, inden man kører Virtual Ribosome er det standardindstillingerne, der bliver brugt til translationen. Disse er følgende:
For at starte translationen skal man trykke på Submit query.
Et output for Virtual Ribosome kan ses i figur 37. Som det fremgår af figuren kan man se både DNA- og proteinsekvenser, hvor proteinsekvensen er den øverste. I eksemplet i figur 37 er der kun benyttet én læseramme. Hvis der havde været benyttet flere læserammer ville disse være ”stablet” oven på hinanden.De tre læserammer, der tilhører den positive DNA-streng, ville således være over DNA-sekvensen, og de tre, der tilhører den negative DNA-streng, ville være under DNA-sekvensen i baglæns aminosyrerækkefølge.
Hvis man vil gemme den translaterede proteinsekvens, kan man højreklikke på FASTA og trykke “Gem som”. Proteinsekvensen bliver da gemt i FASTA-format.
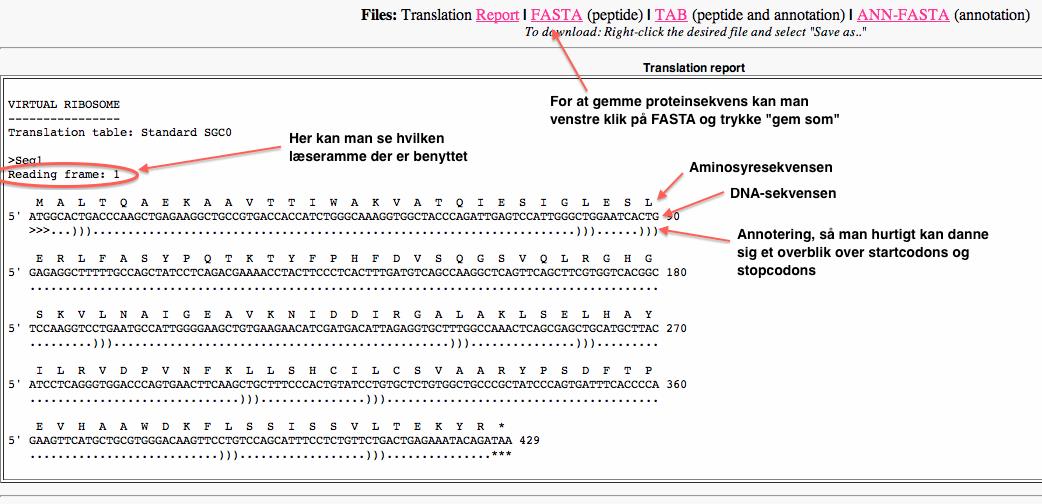
Figur 37. Eksempel på et output fra Virtual Ribosome.