Hvis man kender sammensætningen af nukleotider eller aminosyrer for henholdsvis DNA- eller proteinsekvenser, kan man sammenligne to eller flere sekvenser for at se, hvor meget de ligner hinanden. Sammenligningen af sekvenser foretages DNA mod DNA og protein mod protein, da man ser direkte på, hvilke nukelotider eller aminosyrer, som er identiske i sekvenserne. På engelsk betegnes sekvenssammenligninger som alignments, og at sammenligne sekvenser betegnes ataligne to sekvenser. Termerne alignment og at aligne vil blive brugt i denne teoridel.
Alignments foretages for at se, om to sekvenser er beslægtede. Hvis sekvenserne er beslægtede, er der en sandsynlighed for, at proteinerne har samme funktion, og gennem alignments kan man således bestemme funktionen af ukendte sekvenser. På grund af mutationer i form af indsættelser (eng. insertions) eller fjernelser (eng.deletions) af aminosyrer, kan beslægtede sekvenserne være blevet ændret med tiden, og aminosyrerne kan derfor variere, eller hele dele af sekvensen kan være fjernet.
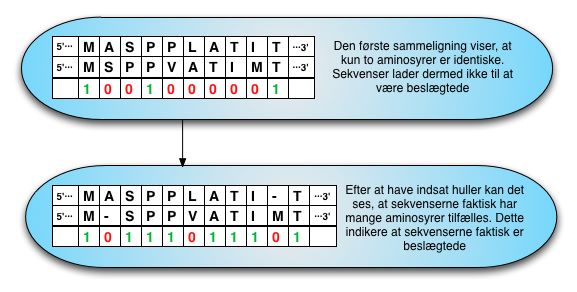
Figur 13. To sekvenssammenligninger. Den øverste er den “originale” uden huller og den nederste er der indsat huller. Et “1” indikerer, at aminosyrerne er identiske, og et “0” indikerer, at der er enten et mis-match eller et hul.
På trods af ændringer i sekvensen kan den overordnede funktion stadig være bevaret, da det er specifikke domæner (f.eks. katalyserende domæner i enzymer, der har betydning for proteinets funktion. Således gør det ikke noget, at mindre betydningsfulde positioner i proteinet er muterede.
På grund af indsættelser eller fjernelser af aminosyrer er de sekvenser, man aligner, ikke altid lige lange. For at tage højde for dette i alignmentet, kan der indsættes huller (eng. gaps) for at indikere, at en eller flere aminosyrer mangler. Ved at indsætte huller kan man opnå et bedre alignment, hvilket fremgår af figur 13.
I teorien kan alle sekvenser alignes, og hvis man indsætter tilstrækkelig mange huller, vil sekvenserne blive lige lange. For at skelne mellem gode og dårlige alignments, dvs. alignments, hvor proteinerne faktisk er beslægtede og alignments, hvor de ikke er, benytter man et scoringssystem.
Scoringssystemet er konstrueret således, at hver alignment får tildelt en score (alignment scoren), som på baggrund af antal identiske aminosyrer (match), antal huller og antal næsten identiske aminosyrer (mis-match, f.eks. position 6, L mod V, i nederste del af figur 13) tildeler alignmentet en score.
Jo højere score, desto bedre sammenligning, da match vil tælle positivt, mens huller/mis-match tæller negativt.
Hvis det antages, at et match scorer 1, et mis-match scorer -1, og huller scorer -2 vil scoren for de to alignments i figur 13 være.
| Sammenligning uden huller: | 1-1-1+1-1-1-1-1-1+1 | = -4 |
| Sammenligning med huller: | 1-2+1+1+1-1+1+1+1-2+1 | = 3 |
Det kan således ses, at selvom der bliver indsat huller, der vægter negativt vil alignmentet alligevel være bedre med end uden huller. Omvendt ville et overtal af huller dominere med en negativ score, og dermed vil et alignment med mange huller ikke være favorabelt.
Som nævnt kan man lave alignments mellem to eller flere sekvenser. Hvis man aligner mere end to sekvenser, kaldes det et multiple alignment. Når man laver multiple alignments, kan man undersøge, hvor tæt beslægtede et stort antal proteiner er. Man kan bruge disse alignments til at lave fylogenetiske træer, således at slægtskabet mellem proteinerne kan visualiseres.
Nogle proteiner er mere identiske på tværs af arter end andre. Ofte er disse proteiner essentielle proteiner, og de indgår i vigtige biologiske processer som metabolisme, proteinsyntese og DNA-replikation. Det skal dog påpeges, at bevarede proteiner ikke altid er essentielle, men det kan antages, at hvis bestemte områder i et protein er bevarede, er disse omrdåder essentielle. Ved at lave multiple alignments af et essentielt protein, der stammer fra forskellige organismer, vil man ud over at se slægtskabet over proteinerne også kunne se slægtskabet mellem organismerne. Dette kan lade sig gøre, da det antages, at forskellen mellem proteinerne relativt set er den samme som forskellen mellem organismerne. Således kan man bruge alignments til at undersøge, hvor beslægtede både proteiner og organismer er.

