Alt om proteiner — UniProt
Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion


Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion
UniProt er en online database, der indeholder informationer omkring proteiner (find det her). UniProt kan anvendes til at indhente viden om et protein, da databasen blandt andet giver et godt overblik over proteinfunktioner. UniProt læres bedst at kende ved at lave søgninger på forskellige proteiner. I øvelse 1b – Aktin, fra mRNA til tredimensionelt protein, øvelse 2a – Myostatins proteininteraktioner og organismers slægtsskab og øvelse 4b – Identifikation og visualisering af ukendt protein arbejder man med UniProt.
I UniProt kan man søge efter proteiner ved at anvende enten accession-nummeret eller ved at søge via fritekst. Hvis man vælger at søge på accession-nummeret, søger man på et specifikt protein og får dermed kun ét resultat. Dette anbefales, hvis man kender accession-nummeret og gerne vil finde informationer om netop det protein. Kender man derimod ikke accession-nummeret, kan man foretage en fritekstsøgning. Hvis man vil søge efter eksempelvis insulin, indtaster man det i søgefeltet (se figur 33) og får dermed en resultatliste med alle de sider fra UniProt, der indeholder tekst, hvori insulin indgår. Da UniProt er koblet sammen med mange af de mest omfangsrige databaser, vil en uspecificeret søgning (som en fritekstsøgning ofte er) i UniProt for det meste give en meget stor mængde af resultater.
Figur 33. Startsiden i UniProt, februar 2020.
For at mindske antallet af resultater kan man gøre søgningen mere specifik. Søgningen kan foretages ved at angive forskellige parametre, blandt andet proteinnavnet, hvilken organisme detstammer fra, den subcellulære lokation etc. Valget af søgningsparametre afhænger af det protein, man vil finde, og hvor nøjagtigt man ønsker, at resultatet skal være.
De nævnte parametre kan bestemmes ved at lave en avanceret søgning gennem Advanced Search (se figur 33). Hvis man eksempelvis vil søge efter Immunoglobulin G fra en mus, kan man i første søgefelt skrive IgG (se figur 34), trykke Advanced Search, dernæst vælge AND og så specificere i det fremkomne felt at resultat OGSÅ skal indeholde det næste søgekriterium. I feltet Field kan man vælge organisme og til slut skrive mus musculus (der er det latinske navn for en almindelig husmus) i det andet søgefelt, se figur 34. NB! Husk at søge på engelske navne eller latinske navne.
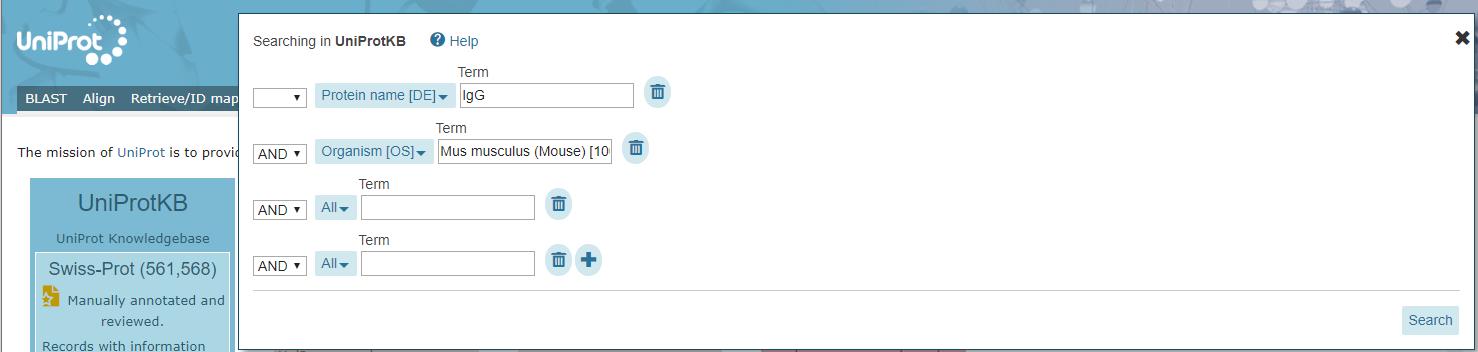
Figur 34. Brugen af en avanceret søgning, Advanced Search.
Søgning med UniProt giver som nævnt ofte mange resultater, så det anbefales at specificere sin søgning medAdvanced Search. Eksempelvis vil søgning på insulin også give resultater med bl.a. insulinreceptor. For at undgå resultater der er beslægtede med det protein, man vil finde, men ikke er selve proteinet, kan man benytte NOT-kriterier. NOT bruges på samme måde som AND, men vil specificere at søgningen IKKE må give resultater, der indeholder det efterfølgende søgekriterium. På denne måde indsnævrer man antallet af resultater og letter det efterfølgende arbejde, der er fortolkningen af søgeresultaterne.
Et eksempel på et søgeresultat kan ses i figur 35. Felter, der er vigtige i forhold til at kunne forstå selve resultatlisten, er markeret med en ring. Informationerne i felterne under punktet fortæller om det givne resultat og giver dermed en indikation af, om man er på rette spor i sin søgning. Felterne inkluderer blandt andet proteinnavnet og navnet på den organisme, proteinet stammer fra. Under feltet Accession findes proteinets accession-nummer, som man kan klikke på for at komme til proteinets UniProt-side.
For at læse om et protein og få informationer om det, skal man gå til proteinets UniProt-side. Dette gøres ved at klikke på accession-nummeret, der står med blåt (se figur 35).
UniProt-siden indeholder en beskrivelse af proteinet. Detaljeringsgraden varierer alt afhængig af den viden, der er opnået om proteinet. UniProt-siden er inddelt i forskellige områder, hvoraf de vigtigste er følgende:
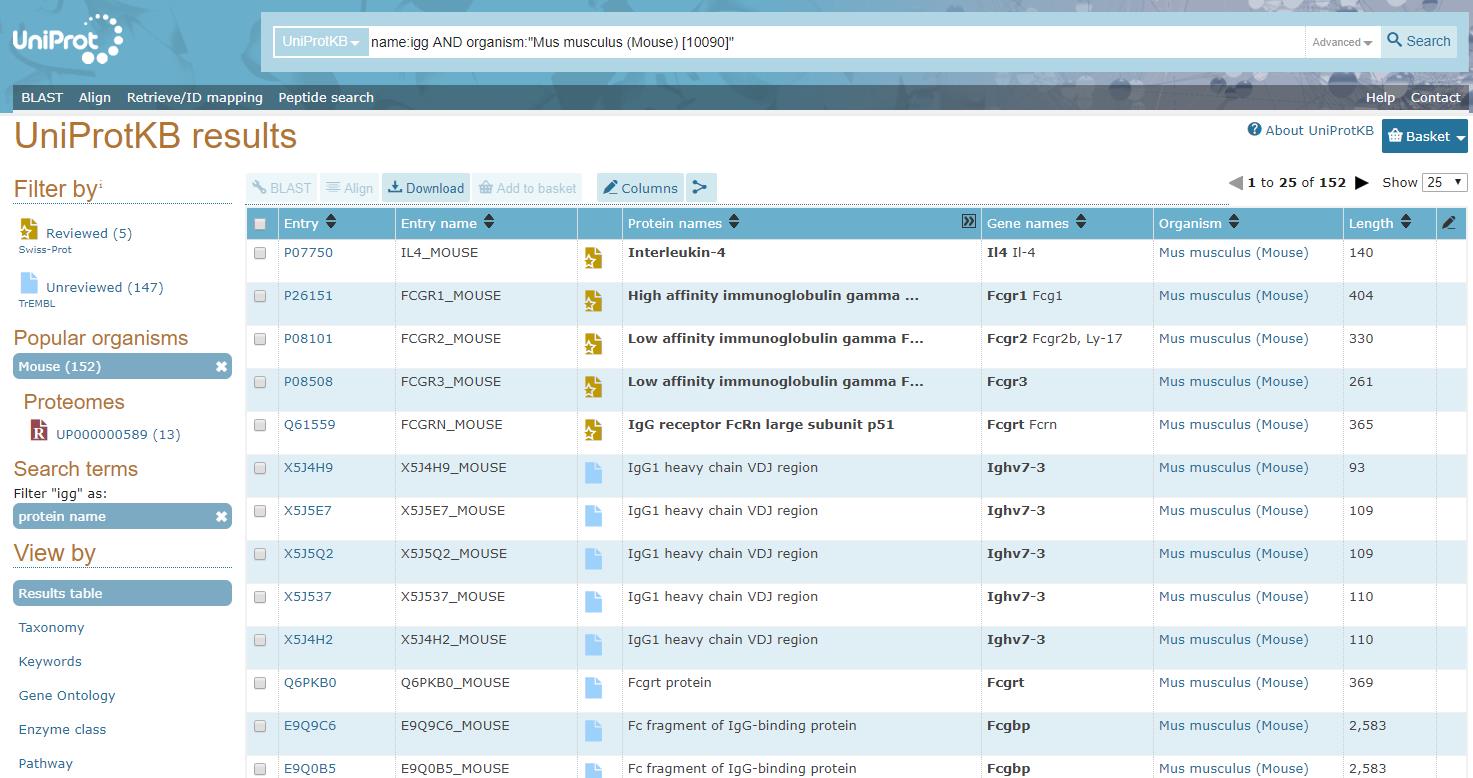
Figur 35. En resultatside efter søgning i UniProt.