Kend dit proteins struktur og funktion — PDB (Protein Data Bank)
Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion


Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion
Protein Data Bank (PDB) er en database, der indeholder proteiner og deres kendte tredimensionelle struktur (find den her). I øvelse 1c – Aktin, fra mRNA til tredimensionelt protein, øvelse 2b – Myostatins proteininteraktioner og organismers slægtsskab, øvelse 3b – Bioinformatisk analyse af antistoffer og øvelse 4c – Identifikation og visualisering af ukendt protein arbejder man med brugen af PDB.
For at et protein kan blive tilgængeligt i PDB, skal det være sekventeret, dvs. man skal kende proteinets aminosyresammensætning og dermed både dets primær-, sekundær- og tertiærstruktur. Proteiner, der er tilgængelige gennem PDB, er ofte i interaktion med andre molekyler, f.eks. metalioner, DNA eller andre proteiner da interaktionerne stabiliserer proteinet.
En af metoderne, hvorpå den tredimensionelle struktur bliver, fundet er ved røntgenkrystallografi. I denne metoder sender man røntgenstråler ind på atomerne, i en struktur og da røntgenstrålen vil blive afbøjet forskelligt alt afhængig af atomernes placering, kan man ved at måle afbøjningen bestemmer strukturen.
PDB-databasen er kædet sammen med en række andre databaser og programmer, blandt andet NCBI og PyMol. Ved at søge på et protein på NCBI kan man undersøge, om proteinet har en kendt tredimensionel struktur. En sådan undersøgelse er mulig, fordi hvert protein i PDB har et PDB-id. Et PDB-id’et er et unikt nummer i stil med et accession-nummer.
PDB id’et er fire karakterer lang og består af både tal og bogstaver f.eks. 1MGY. PDP kan bruges til at:
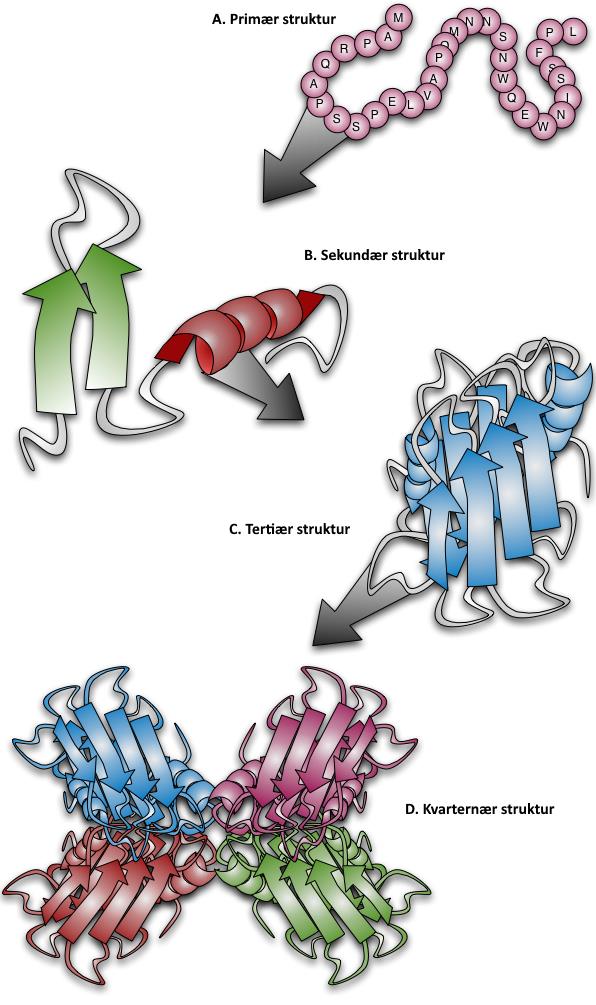
Figur 25. Sammenhæng mellem proteiners strukturformer. I PDB kan man finde tertiær- og kvarternærstrukturer for proteiner.
Den mest simple søgning i PDB-databasen gøres ved at søge på et PDB-id, f.eks. 1JKL. Ved at søge på et PDB-id finder man kun det pågældende protein. En mere kompleks søgning udføres med fritekst, f.eks. ved at søge på “human insulin”. Fritekstsøgning anvendes, hvis man vil undersøge, om PDB indeholder kendte strukturer inden for søgekriteriet, f.eks. om PDB indeholder strukturer af humant insulin.
En ulempe ved fritekstsøgning er det store antal resultater, der ofte fås. Det anbefales derfor at søge direkte på PDB-id’et, hvis man kender det.
På figur 26 kan man se hvor søgningsfelterne er placeret i PDB, og figur 27 viser opbygningen af en resultat side efter endt søgning.
Når den rette struktur er fundet, kan man gå til strukturens PDB-side. Øverst på siden er en række faner (1 på figur 28), der kategoriserer strukturens information. I dette materiale skal fanerne Summary og Sequence benyttes.
I fanen Sequence (se figur 29), er der mere specifikke informationer om strukturen.
Som nævnt kan et protein bestå af flere kæder (subunits). Kæderne kan være både identiske og forskellige, og på Sequence-siden kan man se, hvilke der er identiske og forskellige. Dette kan findes under 1 i figur 29, hvor der også står, hvor mange kæder der er. Man kan vælge, om man vil se informationerne for alle kæderne i strukturen, eller kun dem der er unikke (dvs. at der kun vises en af hver kæde, og identiske kæder bliver dermed ekskluderet), se 2 på figur 29. For hver kæde kan man endvidere se sekundærstrukturen for forskellige dele af sekvensenen og dermed se ved hvilke aminosyrer, de forskellige sekundærstrukturer starter.
Hvis man ønsker at arbejde videre med proteinet i f.eks. PyMol, kan man downloade PDB-filen. Dette gøres ved at trykke Download files i det øverste højre hjørne. Når man har trykket på Download files, skal punktet PDB file (Text) vælges, og PDB-filen bliver dermed downloaded direktet til din download mappe. PDB filen kan åbnes i programmet PyMol, hvor du kan visualisere proteinet.
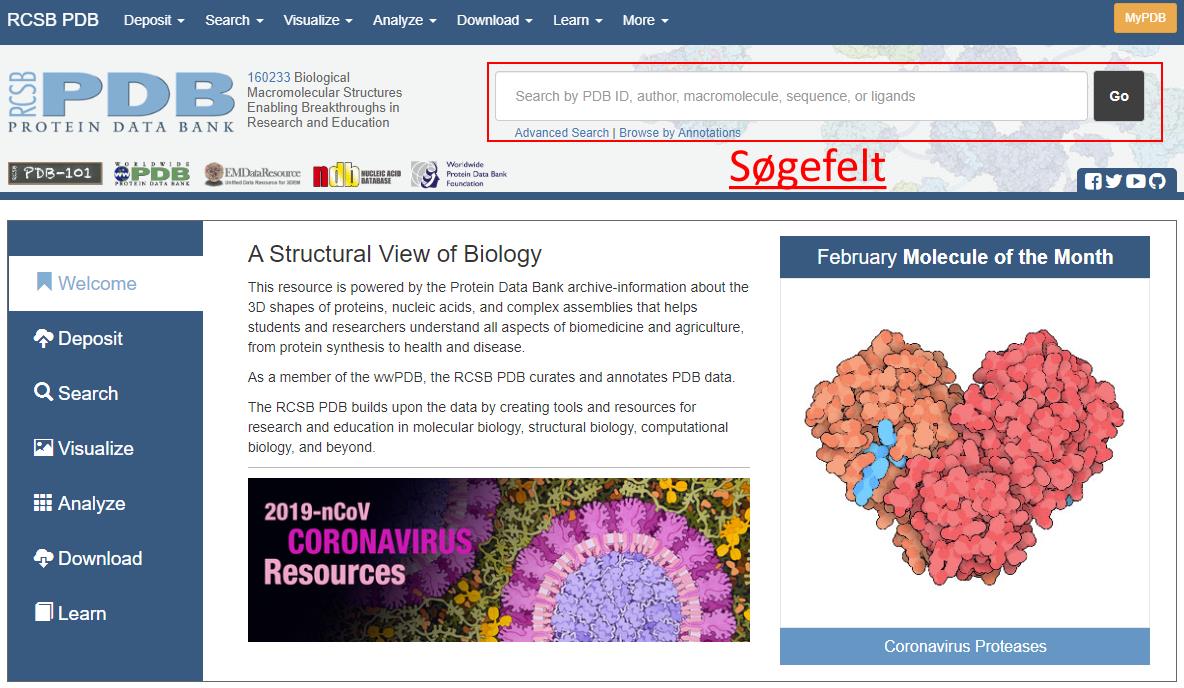
Figur 26. Startsiden for PDB, februar 2020.
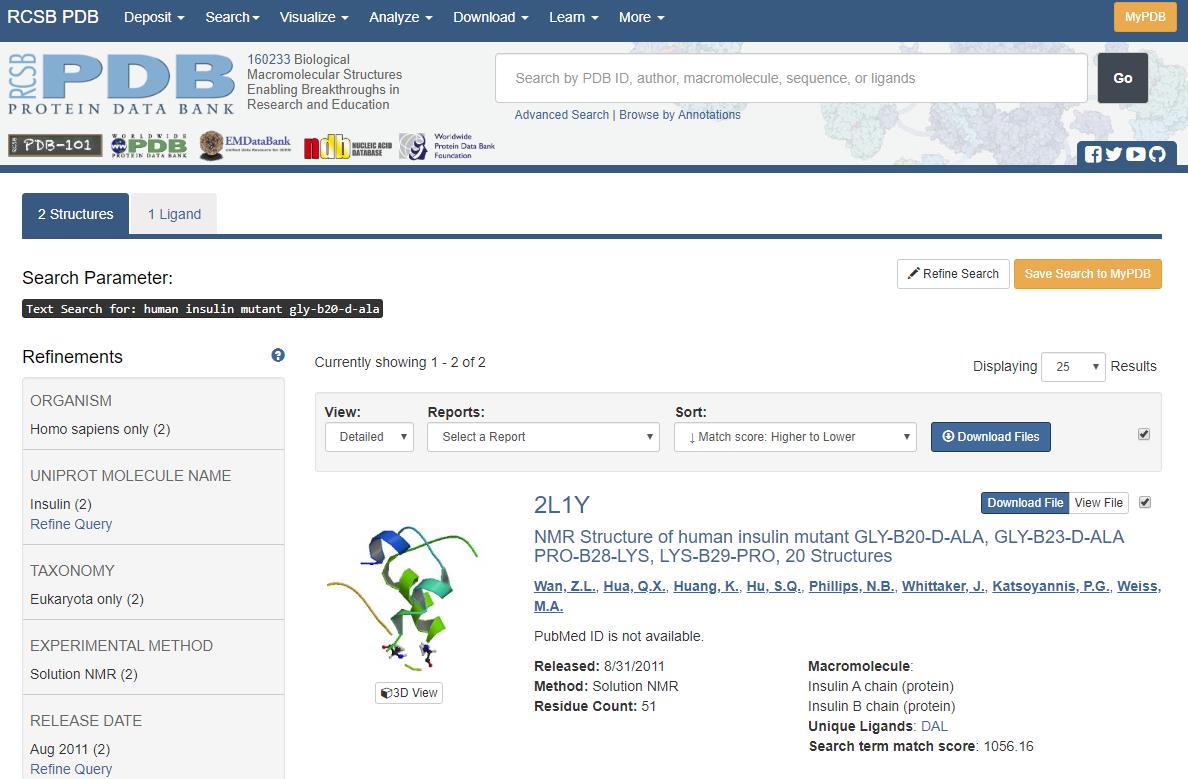
Figur 27. Resultatsiden efter en fritekstsøgning i PDB. Det er den øverste del af siden der af vist, og dermed kun det første af mange resultater. Her er det strukturen med PDB ID 2L1Y, der kan ses.
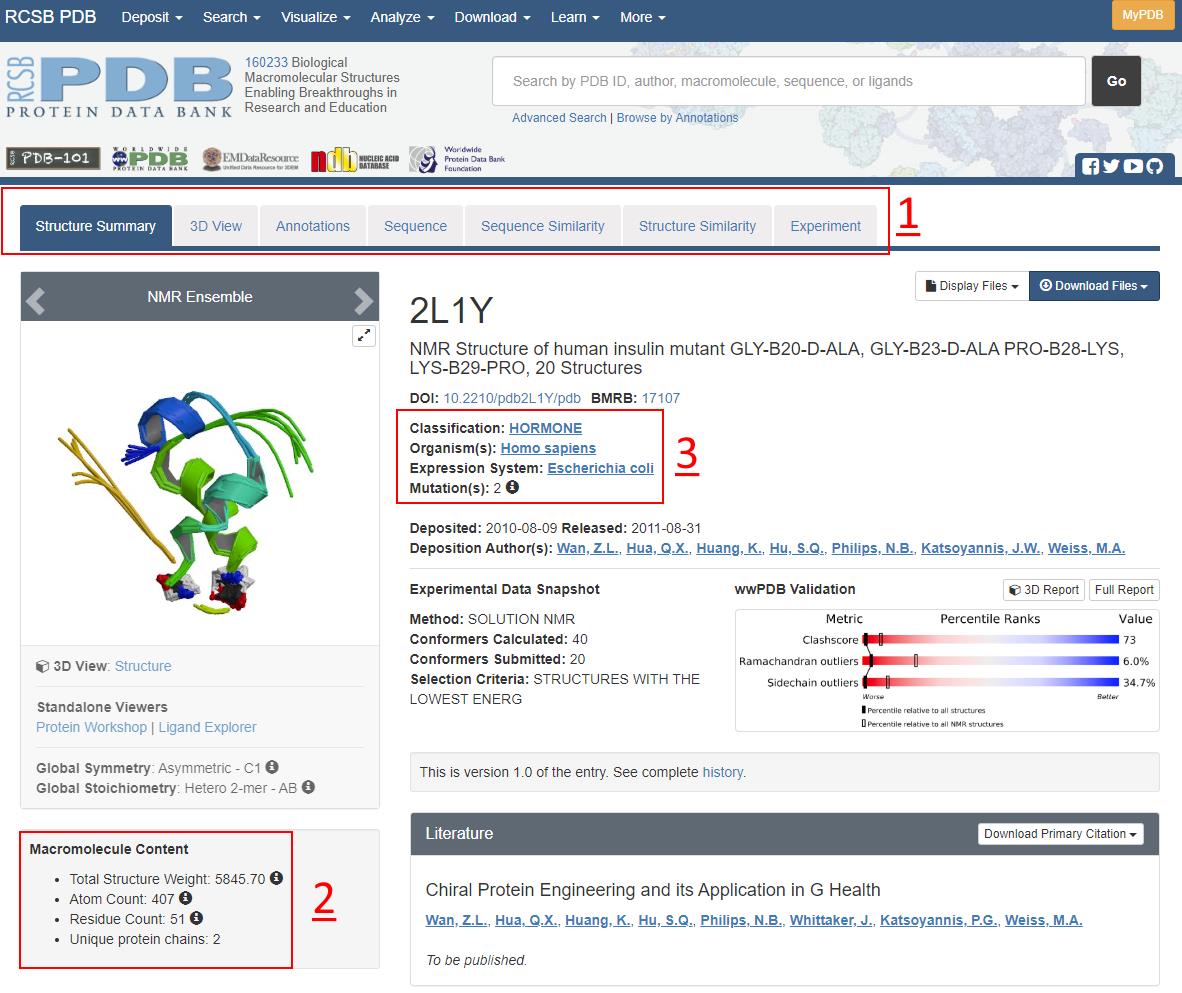
Figur 28. Summary siden for en PDB side. Se brødtekst for detaljer.
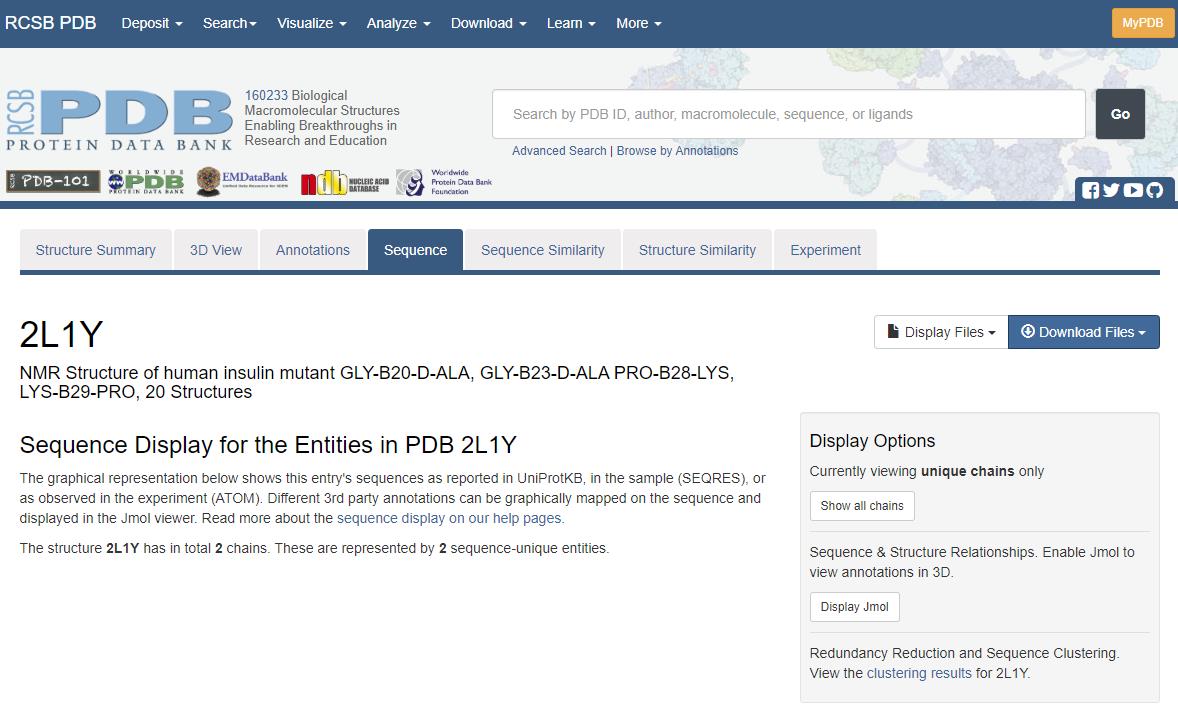
Figur 29. “Sequence” fanen. Siden indeholder information om aminosyresekvensen, samt proteinets sekundære strukturer (se figur 30).
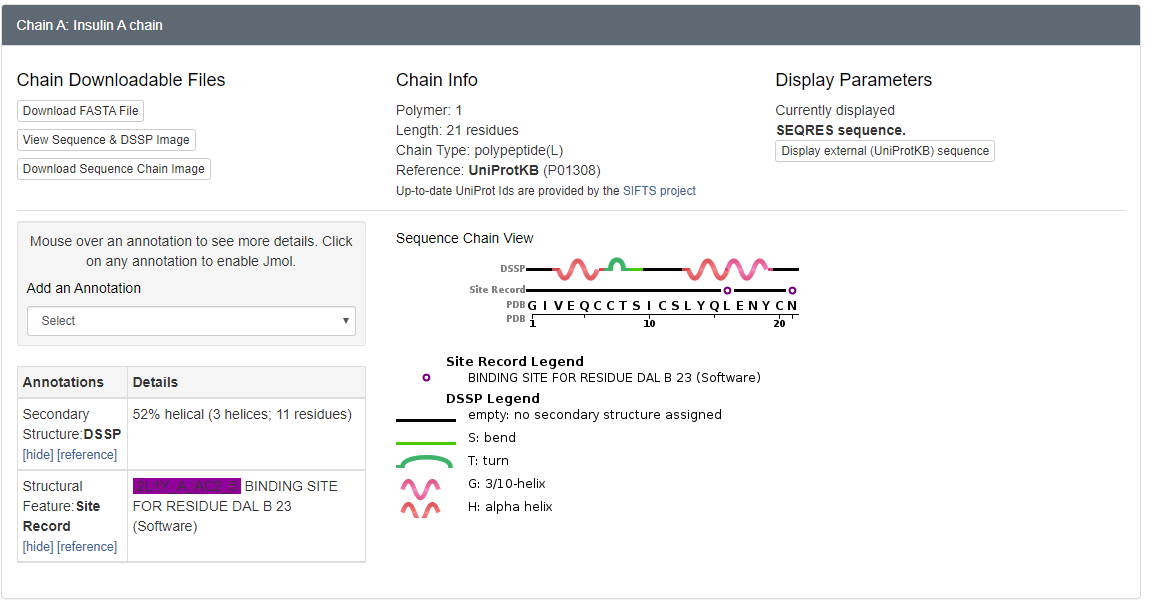
Figur 30. Anden del af “Sequence” siden. Siden indeholder information om hver af kæderne for proteinet, samt hvilke sekundære strukturer der findes, og hvorhenne i aminosyresekven de findes. I figuren er den første kæde af humant insulin vist.