Visualiser proteiner — PyMol
Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion


Denne underside hører til Biotech Academy’s gymnasie projekt Bioinformatik – En introduktion
PyMol er et program, der bruges til at visualisere proteiners tredimensionelle opbygning (find det her). I øvelse 1d – Aktin, fra mRNA til tredimensionelt protein, øvelse 2c – Myostatins proteininteraktioner og organismers slægtsskab, Øvelse 3c – Bioinformatisk analyse af antistoffer og øvelse 4d – Identifikation og visualisering af ukendt protein arbejder man med brugen af PyMol.
PyMol er et værktøj til at undersøge den tredimensionelle opbygning af proteiner. PyMol kan bl.a. bruges til at finde og undersøge bindingsdomæner, katalyserende domæner, interaktioner med f.eks. DNA og meget andet. Et eksempel er visualisering af en transskriptionsfaktor. Her er transkriptionsfaktoren (proteinet) i interaktion med et DNA-molekyle. Ved at visualisere interaktioner kan transkriptionsfaktorens bindingssted på DNA-molekylet bestemmes, og man kan finde de aminosyrer, som er vigtige for bindingen.
Man kan zoome ind på interessante områder, og derved visualisere både sekundær-, men også primærstrukturen.
PyMol er et vigtigt redskab for at forstå enkelte proteiners opbygning og deres interaktioner med andre molekyler.
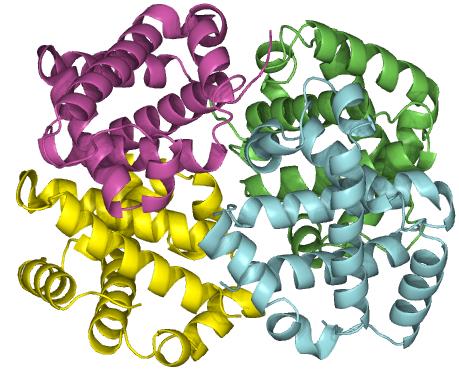
Figur 31. En kvartenærstruktur af et protein, der består af fire forskelligekæder, hver farvet forskelligt. Strukturen er fundet gennem PDB og lavet i PyMol.
Faktaboks: Visualisering af overordnet struktur.
I PyMol kan man se den overordnede struktur af proteinet på forskellige måder. De mest benyttede er:
- Ribbon (dansk: bånd), der en simpel struktur hvor molekylet vises som tynde bånd.
- Sticks (dansk: pinde), der også er en båndstruktur, men her vises sidekæderne også.
- Cartoon (dansk: tegning), hvor man kan se proteiners sekundærstruktur (se infoboks 1).
- Surface (dansk: overflade), som viser overfladen af molekylet. Denne form er god hvis man skal undersøge, hvilke aminosyrer der ligger på overfladen af proteinet og dermed kan interagerer med andre molekyler.
Brugeren af PyMol kan selv bestemme detaljeringsgraden for proteinet. Ønskes få detaljer, kan man gemme forstyrrende elementer, se kommandoen her. Forstyrrende elementer kan f.eks. være sidekæder og hydrogenatomer, da disse er tilstede på hver aminosyre og let kan give et rodet billede.
Hvis der omvendt er dele som sidekæder, atomer etc. man gerne vil undersøge kan de vælges separat. Herefter kan de farves og præsenteres som ønsket, se her for kommandoerne til dette. Ved at præsentere enkelte dele anderledes end hele proteinet kan de nemmere undersøges af brugeren.
PyMol-vinduet, som man arbejder i, er opdelt i flere forskellige dele:
Figur 32 viser et eksempel på et PyMol-vindue, og de forskellige dele PyMol er bygget op af.
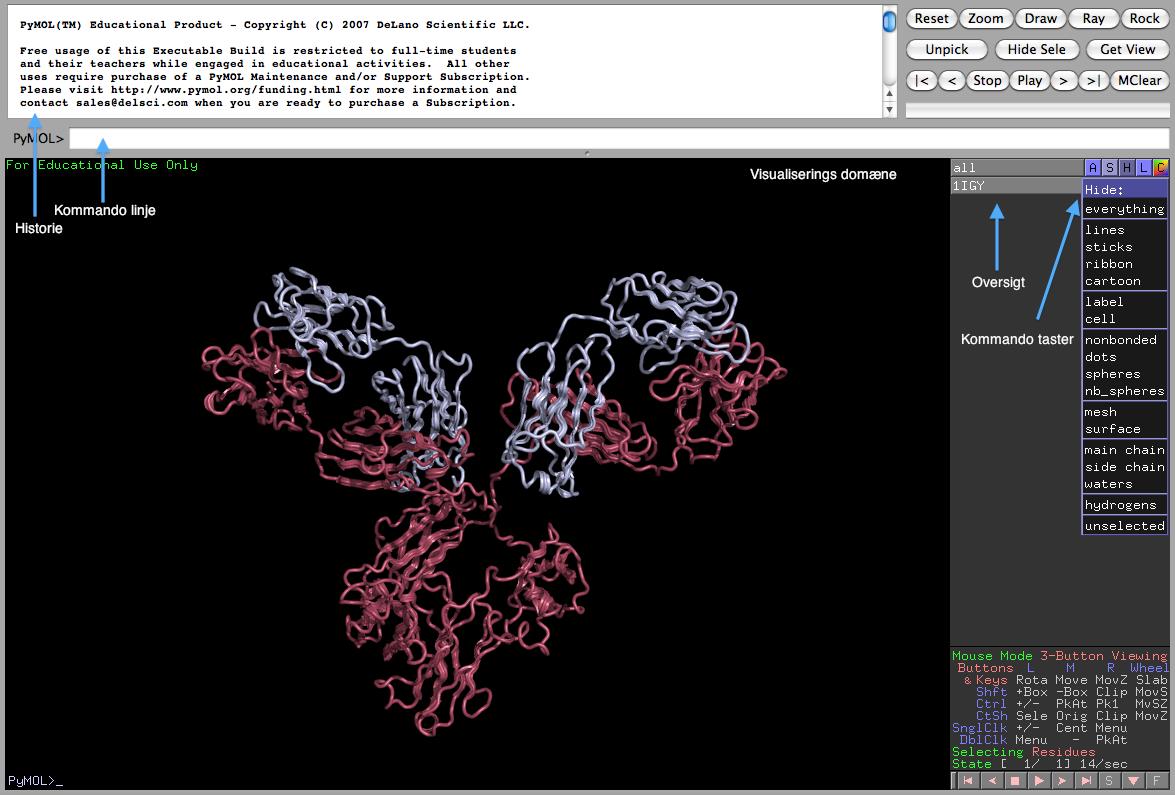
Figur 32. Eksempel på et PyMol vindue.
Som nævnt er PyMol- og PDB-databasen kædet sammen. Hvis den benyttede computer har internetforbindelse, er PyMol i stand til automatisk at downloade det protein, man ønsker at arbejde med. Proteinet downloades direkte fra PDB, hvor man blot skal benytte PDB-id’et til at hente proteinet. Downloading gøres gennem kommandolinjen med kommandoen fetch efterfulgt af proteinets PDB-id.
En anden måde at hente protener til PyMol er ved at downloade selve PDB-filen for det pågældende protein, og derefter vælge open i filer. Når et protein er blevet hentet ind i et PyMol-vindue, vil dets navn, i form af PDB-id’et, blive vist i oversigtsdelen til højre i skærmen (se figur 32).
Ved at benytte ASHLC-tasterne ud for proteinets navn specificeres kommandoer for det protein, der er valgt.
Det er muligt at have flere proteiner i samme PyMol. Det/de proteiner, man ønsker at se, kan markeres ved blot at trykke på proteinets navn i oversigtsdelen.
For at få den bedste visualisering af proteinet er det en god ide at ændre på presentationen af proteinet. En trinvis guide til at visualisere et protein i PyMol kan ses nedenfor, hvor PyMol-kommandoerne også kan vises:
Her er en liste med nyttige kommandoer til PyMol.
Kommandolinje kommandoer:
Hent protein
Hent protein: |
fetch PDB ID |
Find kæder/dele: |
show labels |
Visualiserings strukturer
Vis alt: |
show everything |
|
Gem alt: |
hide everything |
|
Vis båndstruktur: |
show ribbon |
|
Vis tegningsstruktur: |
show cartoon |
|
Vis overfladestruktur: |
show surface |
|
Vælg specifikke dele: |
sele NAVN_PÅ_DELEN, DELENS_PYMOL_NAVN |
Eksempler: |
|
Vælg kæde A: |
sele A, chain A |
Vælg aminosyre 1 til 100: |
sele aminosyre1-100, resi 1-100 |
Vaæl aminosyre 1, 3 og 6: |
sele aminosyre1_3_6, resi 1+3+6 |
Vælg aminosyre 3 på kæde B: |
sele position3, chain B resi 3 |
Farv hele proteinet: |
color FARVEN |
Eksempel: |
color red |
| Farv en specifik kæde: | color FARVEN, KÆDEN |
Eksempel: |
color red, chain A |
Farv en specifik del defineret af brugeren: |
color FARVEN, DEL_NAVNET |
Eksempel: |
color red, position3 |
NB! Inde dele farves, kan delen eventuelt først vælges ved at benytte sele kommandoen.
Sammenligne to proteiner: |
align PROTEIN 1, PROTEIN2 |
Zoom ind på hele proteinet: |
zoom all |
Zoom ind på en specifik del |
zoom NAVN_PÅ_DEL |
Eksempel: |
zoom chain A eller zoom position3 |
Zoom ind på en specifik del med en specifik Angstrøm |
zoom NAVN_PÅ_DEL, ANGSTRØM |
Eksempel: |
zoom chain A, 8 |
ASHLC taster:
Tasterne kan benyttes til at lave de samme kommandoer som ved brug af kommandolinjen. Dog er der visse kommandoer, der er lettere gøres ved at bruge ASHLC tasterne. Dette er f.eks. generel visning/ikke-visning samt farvning af hele proteinet.
Nedenfor er visse eksempler.
| Gem vandmolekyler: | H-tast → hydrogens |
| Vis sidekæder: | S-tast → side chain → sticks |
| Farv ift. den sekundære struktur: | C-tast → by ss → DEN ØNSKEDE FARVE |
| Put mærkater på aminosyre, kæder eller atomer: | L-tast → residue, chain, segment eller atom → name |