CRISPR/Cas9 – Den Genteknologiske Revolution
Resume: CRISPR-teknologien stammer fra prokaryoters immunforsvar, hvor det eksempelvis fungerer som beskyttelse imod virusangreb. CRISPR er en forkortelse af Clustered Regularly Interspaced Short Palindromic Repeats, hvilket beskriver den genomiske struktur af det prokaryote immunforsvar. CRISPR associerede (Cas) proteiner er immunsystemets funktionelle enheder. Den mest anvendte type Cas9 indenfor genteknologi stammer fra bakterien Streptococcus pyogenes.
Alt, der kan defineres som liv, har en enestående ting tilfælles. Alle de informationer, der danner de enkelte organismer, er kemisk lagret i et makromolekyle, som fungerer på den samme grundlæggende måde i mennesket og alle andre organismer. DNA er dét molekyle, der udgør vores genetiske informationer igennem sin genetiske kode. Det har en enorm betydning, at livet deler grundlagene for denne genetiske kode, da det betyder, at det er muligt at tage informationer fra én organisme og bruge dem i anden. Gensplejsning gør det muligt at flytte rundt på gener og styre deres aktivitet, hvormed vi faktisk kan kontrollere livets udformning. Genteknologi kan bruges til at skabe uanede mængder af genetiske kombinationer, der eksempelvis kan give os bedre fødevarer, ændre organsimers egenskaber, bruges til produktion af medicin eller ligefrem kurere sygdomme. Det er kun fantasien og teknologien, der sætter grænser for mulighederne – heldigvis er teknologien kommet langt!
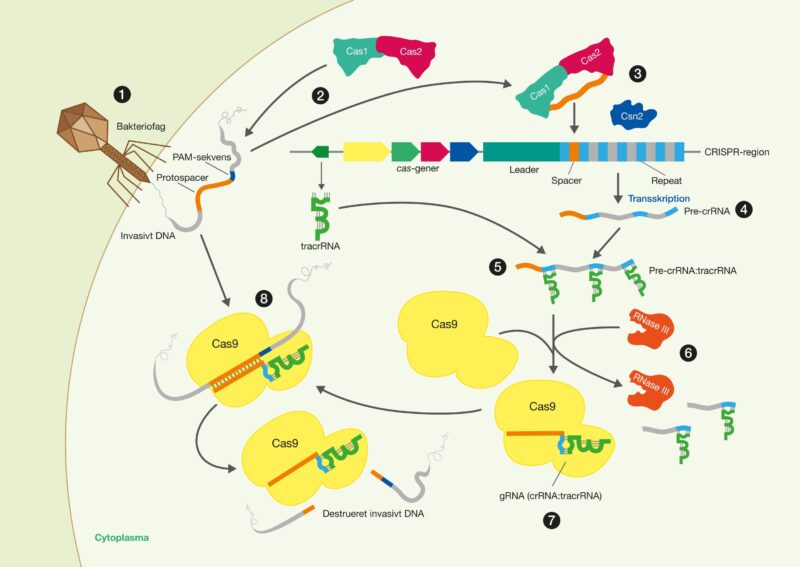
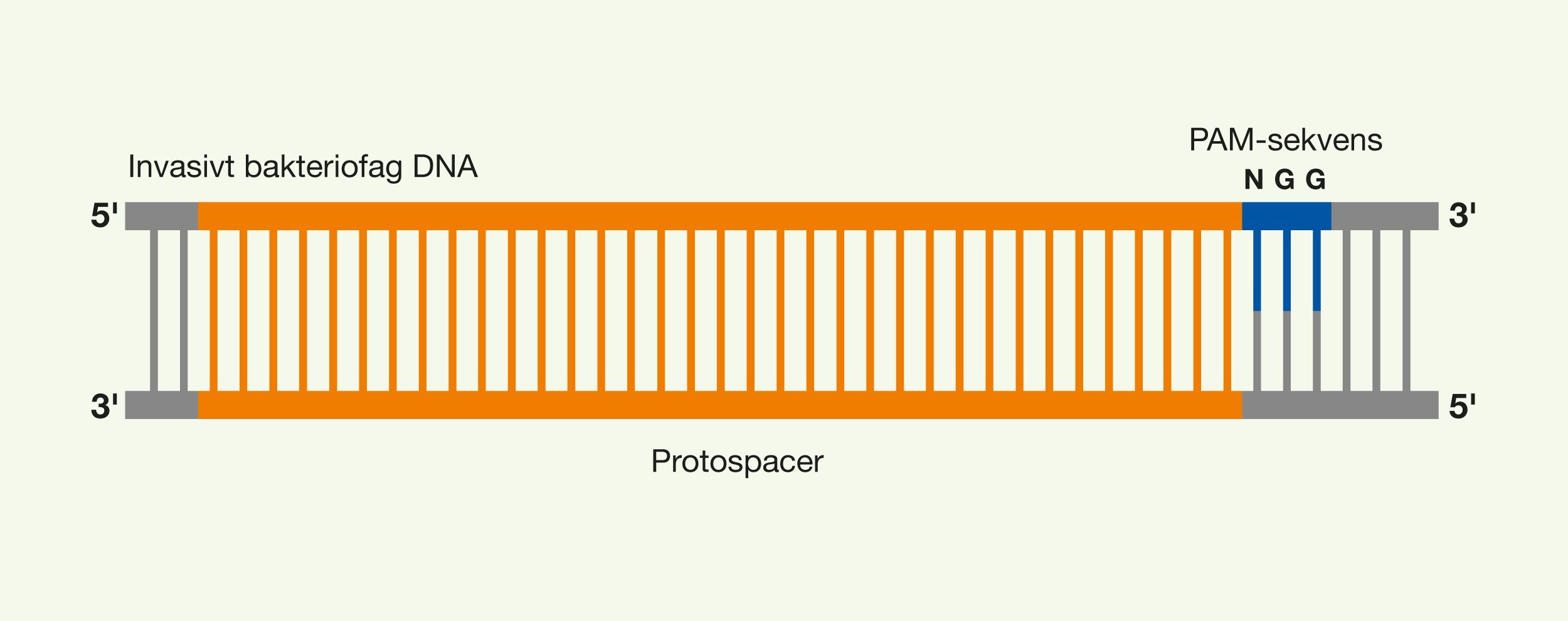
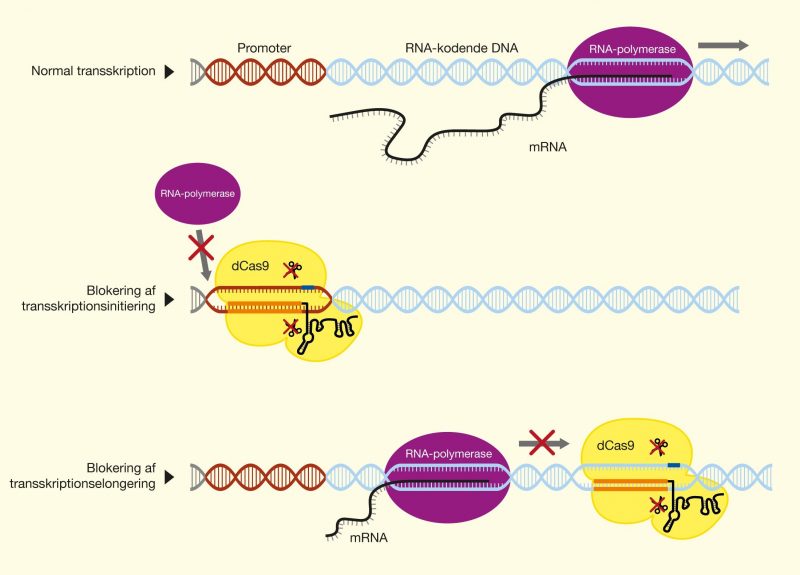
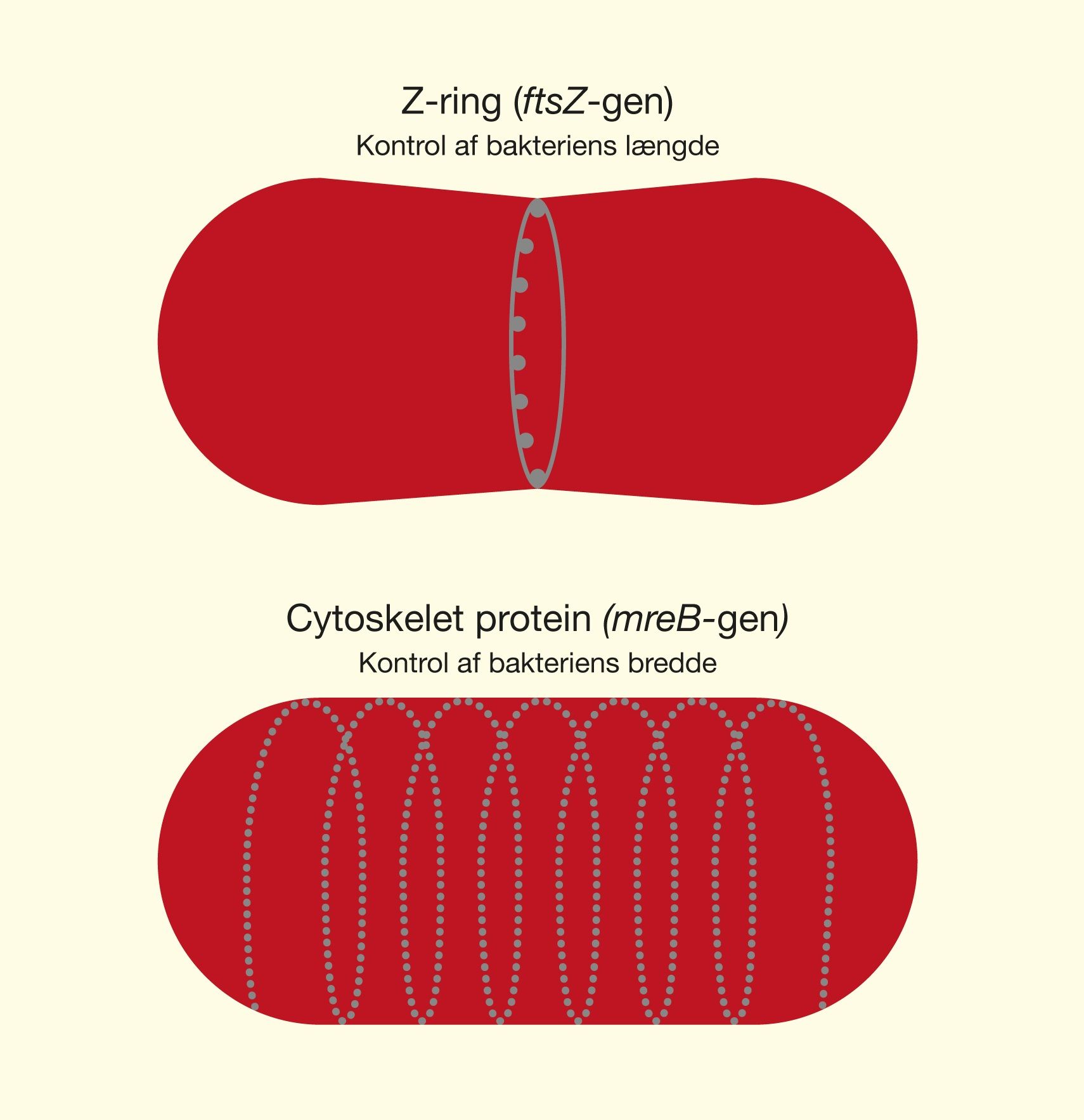
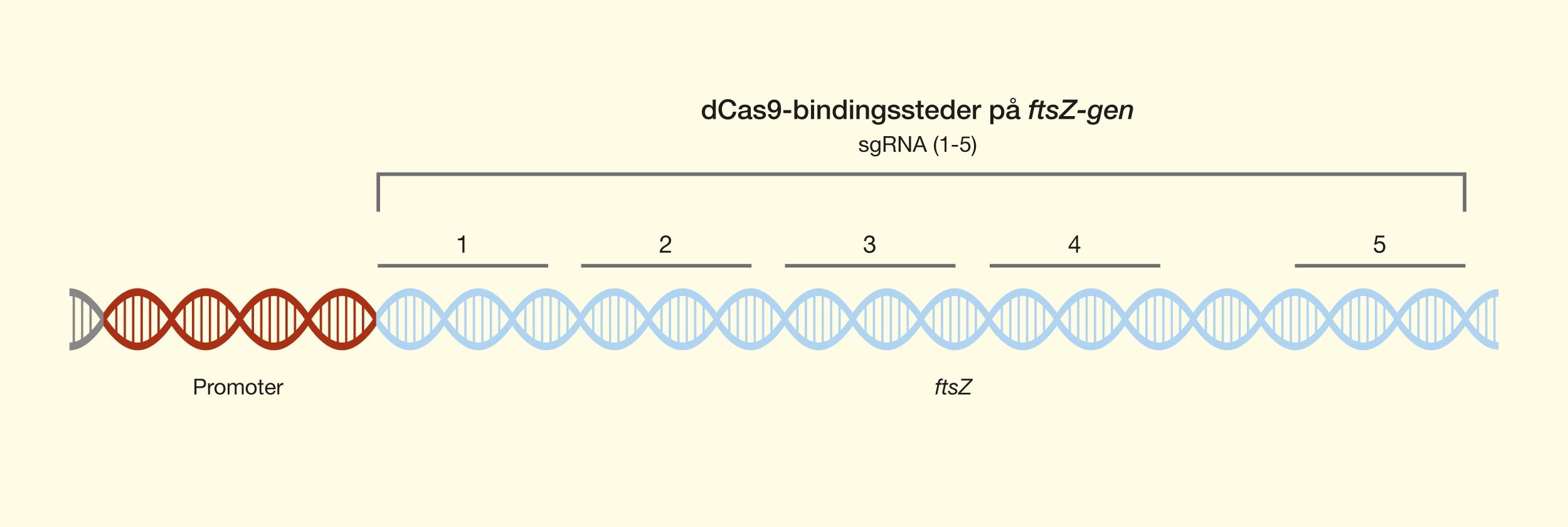
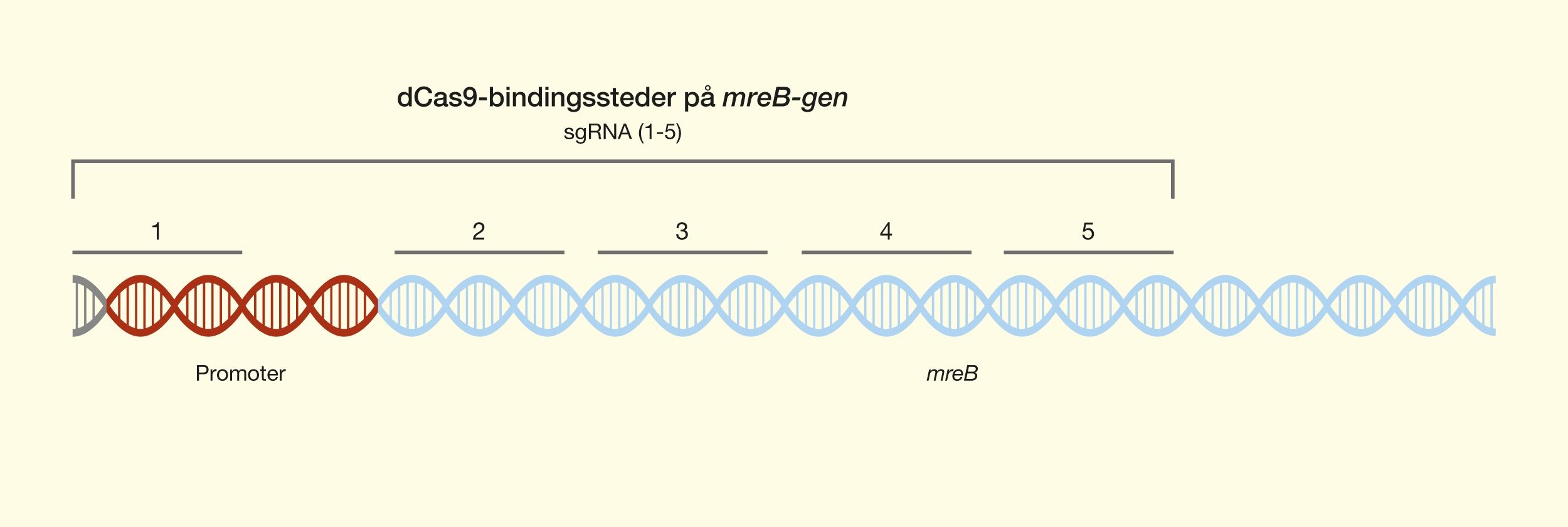
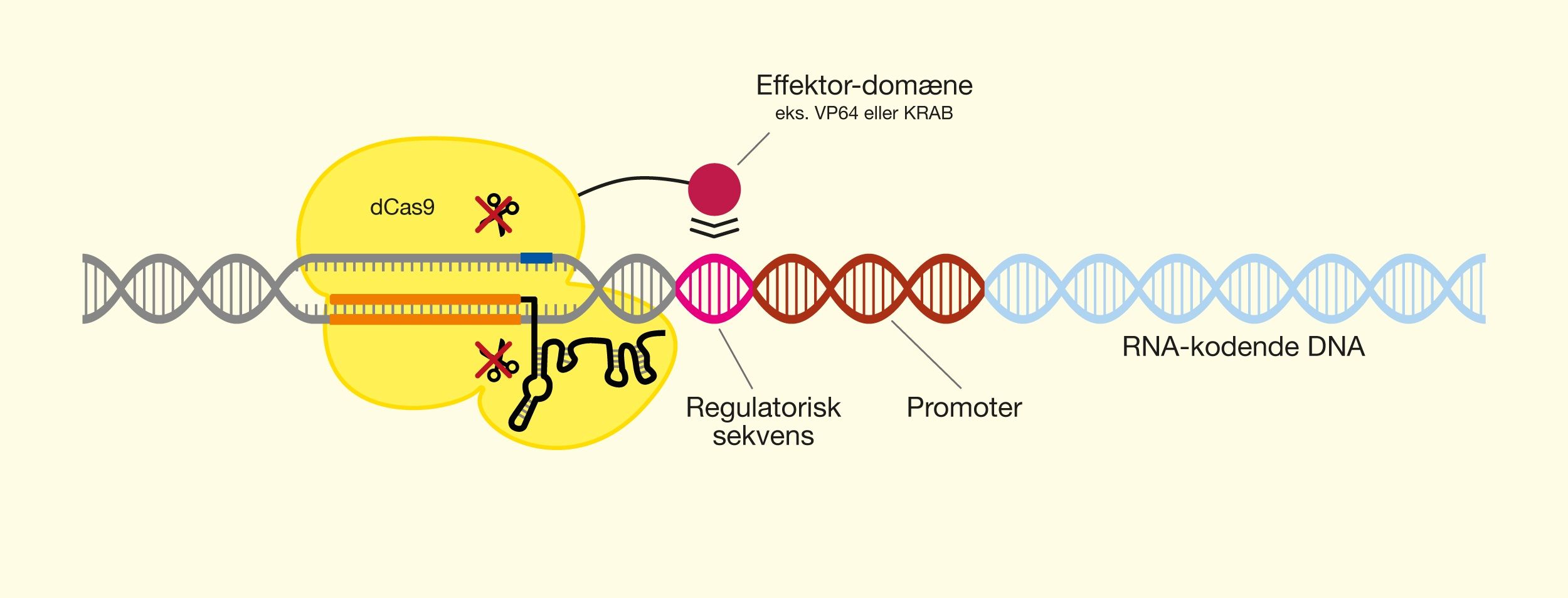
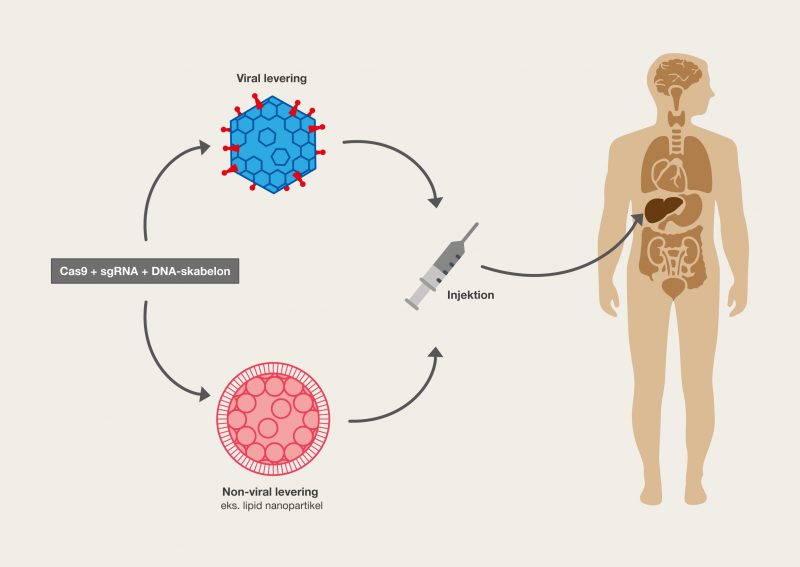
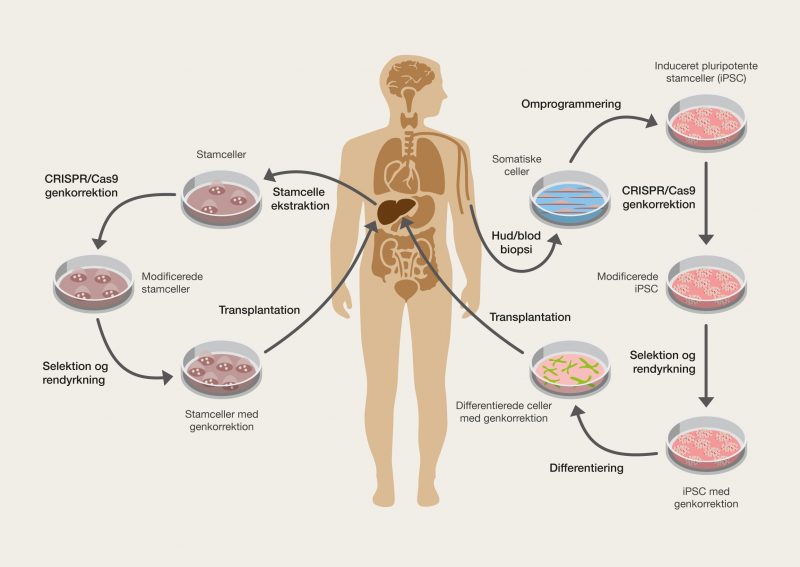
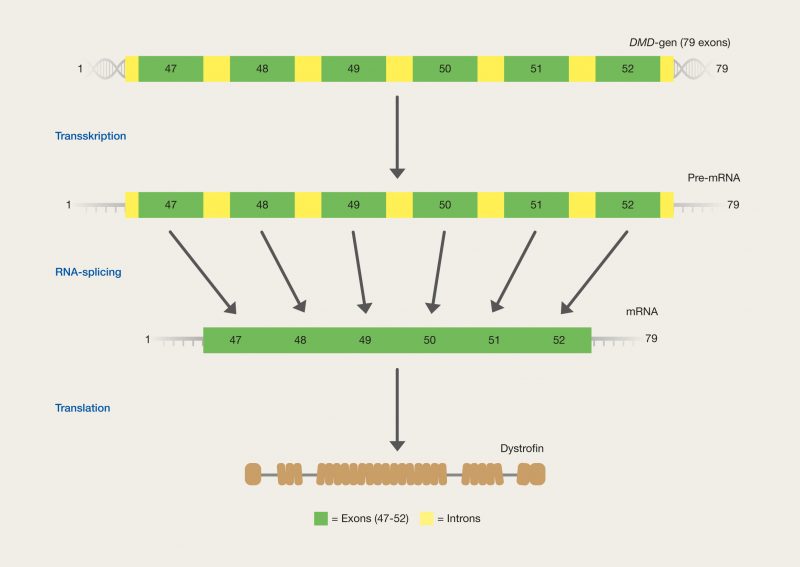
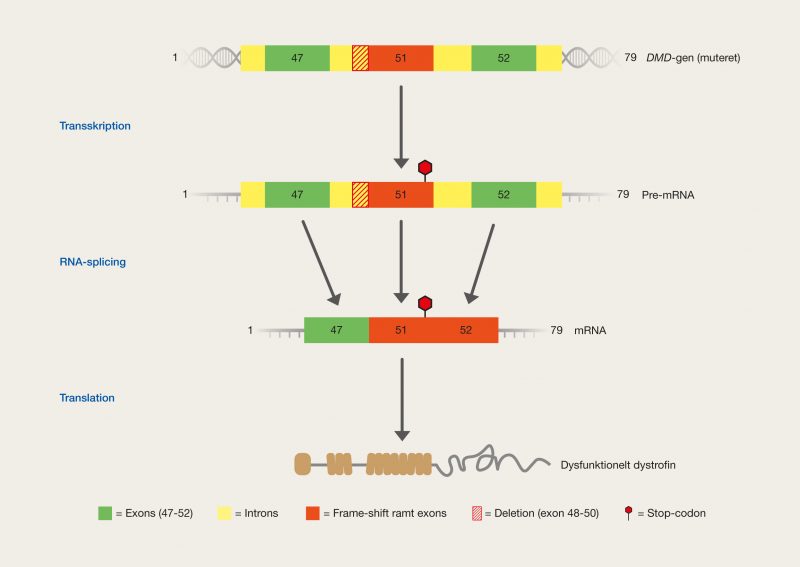
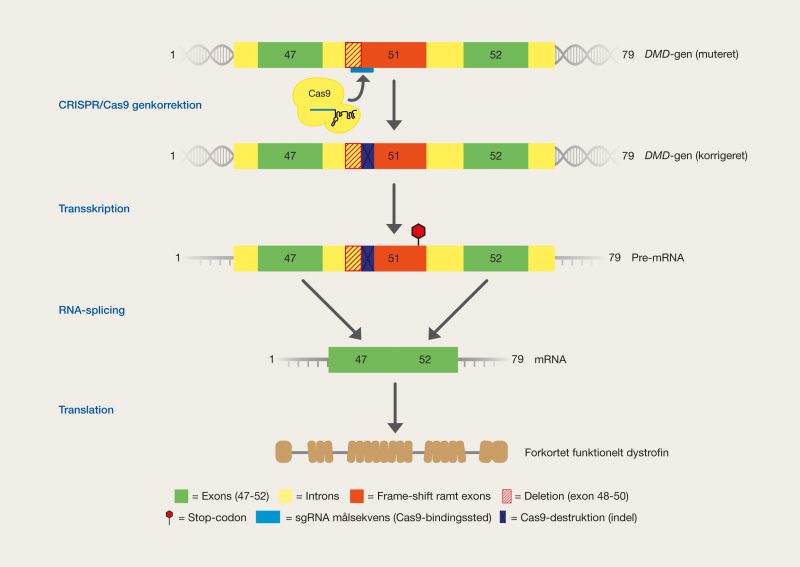
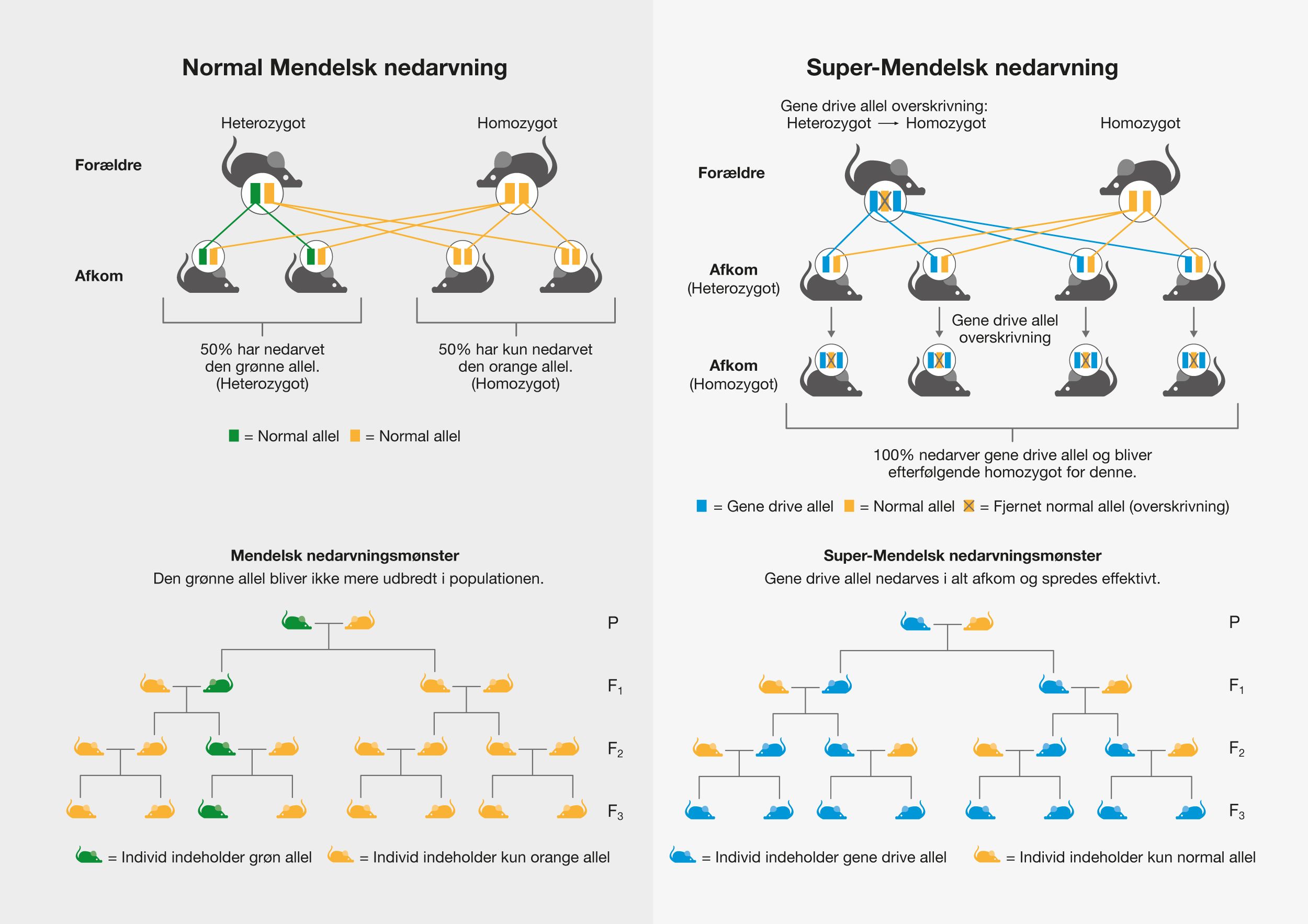
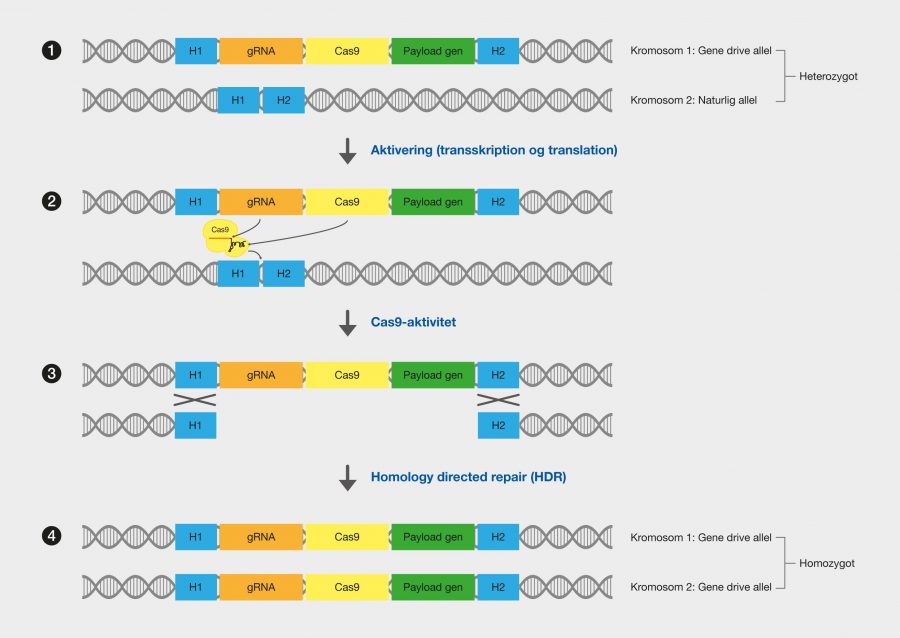
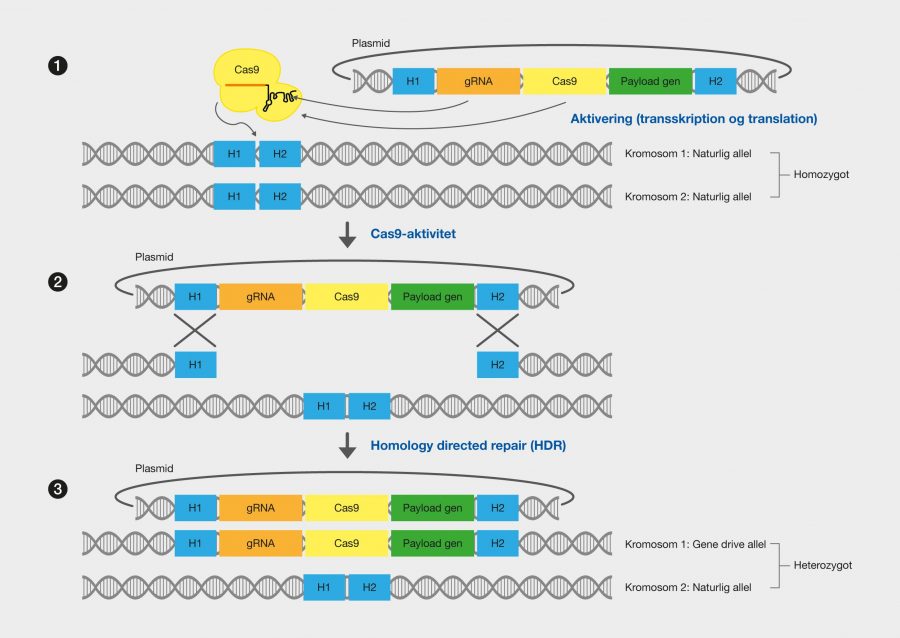
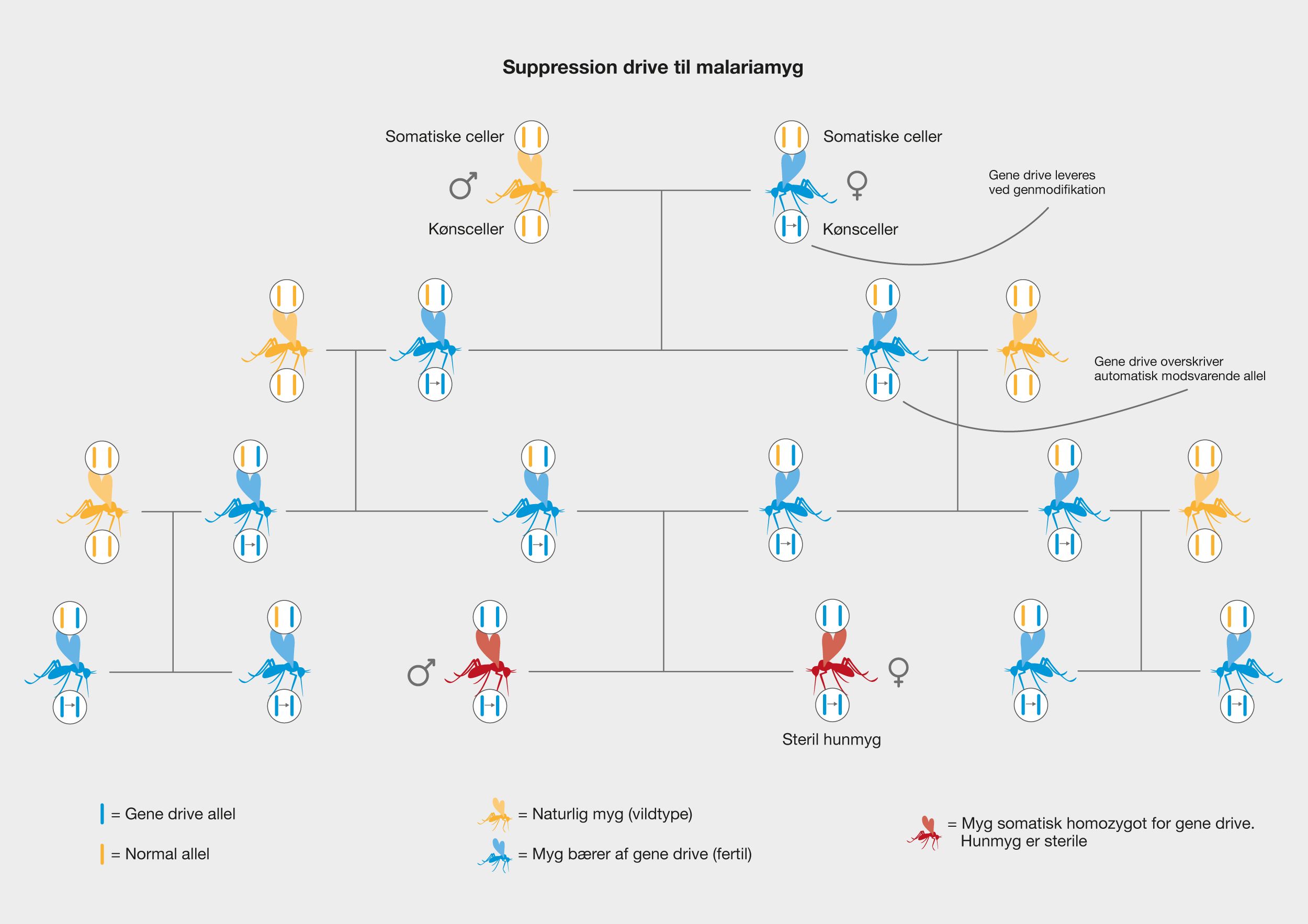
En af de nyeste teknologier er CRISPR/Cas9, der har et stort potentiale, fordi det giver muligheden for at kunne klippe i DNA med enorm præcision og effektivitet, på en lettere og billigere måde. Denne teknologi er baseret på ét protein, kaldet Cas9. Proteinet er styret af et stykke RNA, kaldet guide RNA (gRNA), som giver proteinet dets egenskab til at nøjagtigt identificere DNA-sekvenser, binde sig til dem og klippe dem over, ved at danne præcise dobbeltstrengsbrud i DNA. Grunden til, at Cas9 har så stort potentiale er, at proteinet let kan omprogrammeres til at ramme nye DNA-sekvenser ved udskiftning af dét gRNA, der styrer proteinet. Cas9’s nøjagtige dannelse af dobbeltstrengsbrud tillader eksempelvis indsættelse af DNA-sekvenser i helt bestemte positioner, hvormed man kan opnå nye genfunktioner. Man kan også danne mutationer, der kan deaktivere specifikke gener og dermed fjerne specifikke egenskaber fra organismer.
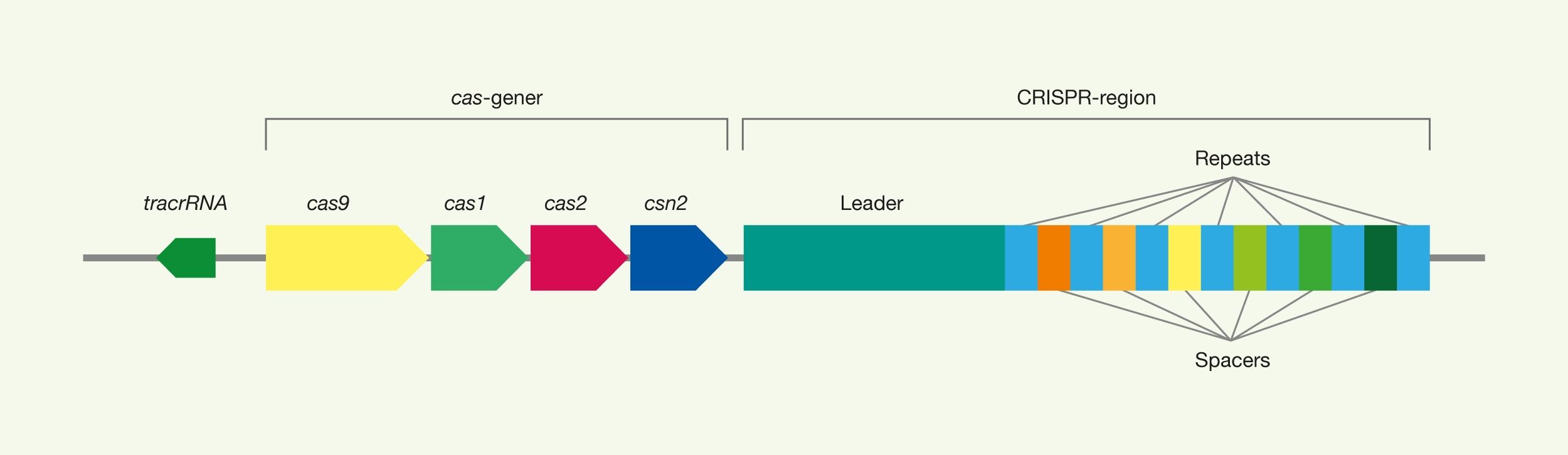
CRISPR-teknologien stammer fra prokaryoters immunforsvar, hvor det eksempelvis fungerer som beskyttelse imod virusangreb. CRISPR er en forkortelse af Clustered Regularly Interspaced Short Palindromic Repeats, hvilket beskriver den genomiske struktur af det prokaryote immunforsvar. CRISPR associerede (Cas) proteiner er immunsystemets funktionelle enheder. Den mest anvendte type Cas9 indenfor genteknologi stammer fra bakterien Streptococcus pyogenes.
CRISPR/Cas9 er et pragteksemplar på et bioteknologisk værktøj, der er udviklet fra naturens egne funktionsmekanismer.
Grundteorien vil give en god grundlæggende forståelse for den molekylære funktion af CRISPR/Cas9 og hvordan vi kan udnytte den til genmodifikation. Herefter kan de forskellige cases læses uafhængigt af hinanden.