Design af sgRNA
Denne underside udgør tredje del af teorien for Biotech Academys materiale om CRISPR-Cas9.


Denne underside udgør tredje del af teorien for Biotech Academys materiale om CRISPR-Cas9.
For at udføre genmodifikation med Cas9 i et gen i en given organisme, må man først designe gRNA, der kan genkende sekvensen. Single chimeric guide RNA (sgRNA) er en type kunstigt gRNA, som man selv kan designe alt efter hvilke DNA-sekvenser, der skal modificeres.
Den mest anvendte type Cas9 stammer fra S. pyogenes immunforsvar. Her bruger Cas9 en type gRNA som består af et kompleks af to RNA-fragmenter. Det kunstigt fremstillede sgRNA er en fusion af disse to RNA-fragmenter. I 5’-enden af sgRNA haves det 20 nukleotider lange segment, der anvendes til at identificere DNA-sekvenser. Disse udvælges blot som komplementære til den DNA-sekvens man vil modificere. Den anden ende af sgRNA har en fast struktur, som tillader at Cas9 kan binde sig til det.
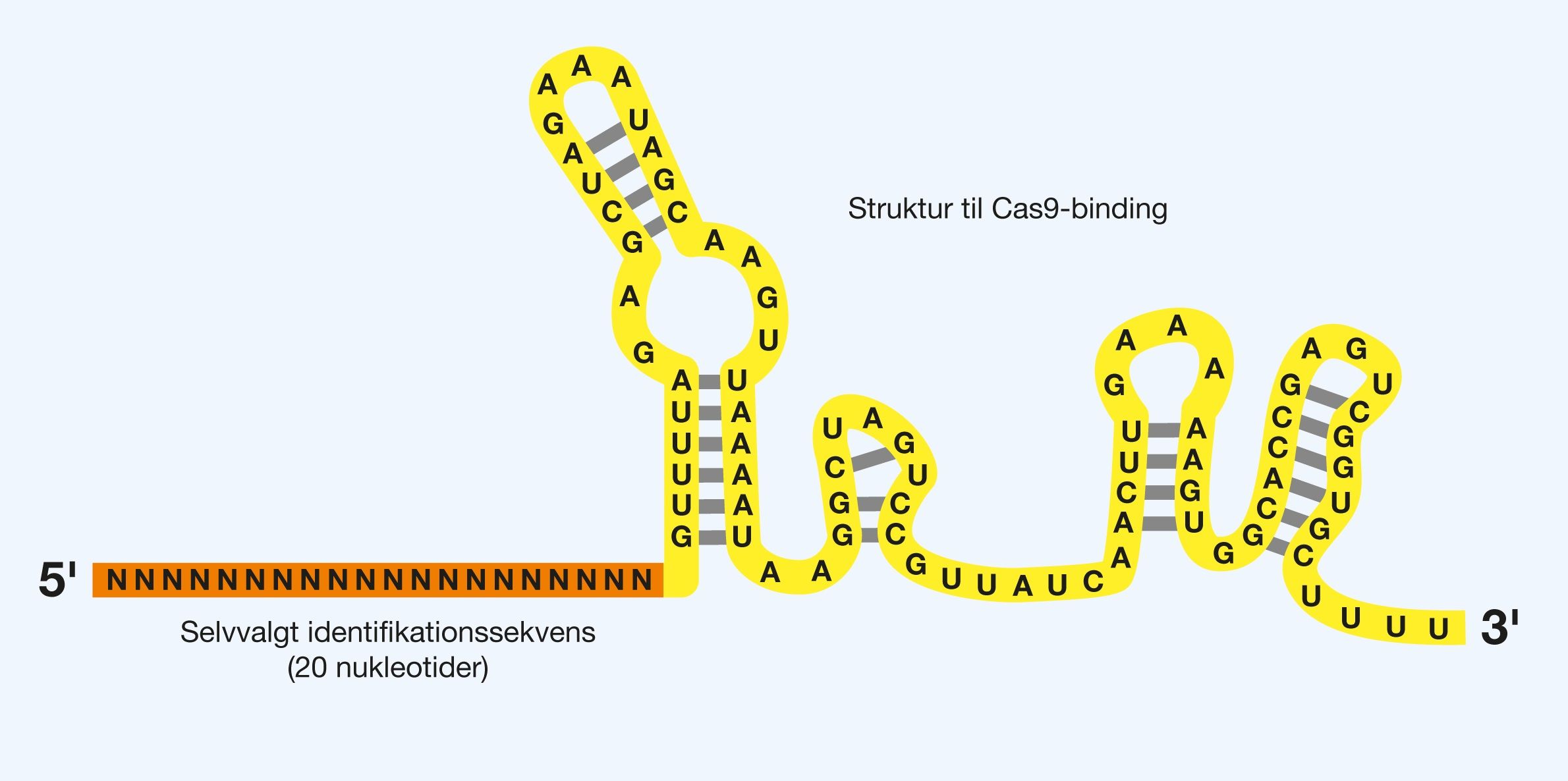
Figur 9. Strukturen af sgRNA, hvor identifikationssekvensen på 20 nukleotider i 5′-enden kan varieres efter eget valg, sådan at forskellige DNA-sekvenser kan eftersøges og blive kløvet af Cas9. ‘N’ er ensbetydende med, at der kan være en enhver nukleotid på positionen. Den faste struktur mod 3′-enden bruges af Cas9 til at binde gRNA.
En strategi for design af sgRNA kunne være:
PUT– DNA-sekvensen ligger i direkte forlængelse af PAM-sekvensens 5’-retning.
PUT– DNA-sekvensen ligger på den modsatte streng af PAM-sekvensen.
Placeringen af de 20 genkendte nukleotider kan ses på figur 4.
Da sekvensen nu er identificeret, skal sekvensen blot oversættes ved brugen af viden om Watson-Crick baseparring. Her husker man, at Adenin (A) og Thymin (T) er et par, og at Cytosin (C) og Guanin (G) er et par. For RNA gælder det at Thymin skiftes ud med Uracil (U), der også danner par med Adenin.
Eksempelvis kunne man have fundet DNA-sekvensen 3’-ACTGGCTAGTACTGCAATGC-5’, denne ville blive oversat til de 20 nukleotider af sgRNA på denne måde:
DNA-sekvens: 3’-ACTGGCTAGTACTGCAATGC-5’
PUT_CHARACTERS_HERECHARACTERS↓
sgRNA-sekvens: 5’-UGACCGAUCAUGACGUUACG-3’
Denne sekvens kan dermed indsættes som de 20 nukleotider i ens sgRNA, angivet med ’N’ i figur 9. Dermed har man designet sit sgRNA færdigt, som nu kan genkende DNA-sekvensen.
Det designede sgRNA kan nu blive fremstillet, hvilket let kan gøres ved at indsende sgRNA-sekvensen til et firma, der specialiserer sig i at lave syntetisk RNA. Der findes flere versioner af gRNA, hvor man har prøvet at ændre længderne af de forskellige segmenter for at optimere effektiviteten og specificiteten for Cas9-identifikationen. Man har før brugt den naturlige form af gRNA bestående af RNA-komplekser til genmodificering, hvilket ikke er lige så effektivt. Heldigvis er det også mere praktisk at lave et helt stykke syntetisk RNA, der er klar til brug, fremfor at skulle rode med produktionen af flere stykker RNA, der først skal danne komplekser med hinanden før brug.