CRISPR/Cas9 komplikationer
Denne underside udgør femte del af teorien for Biotech Academys materiale om CRISPR-Cas9.
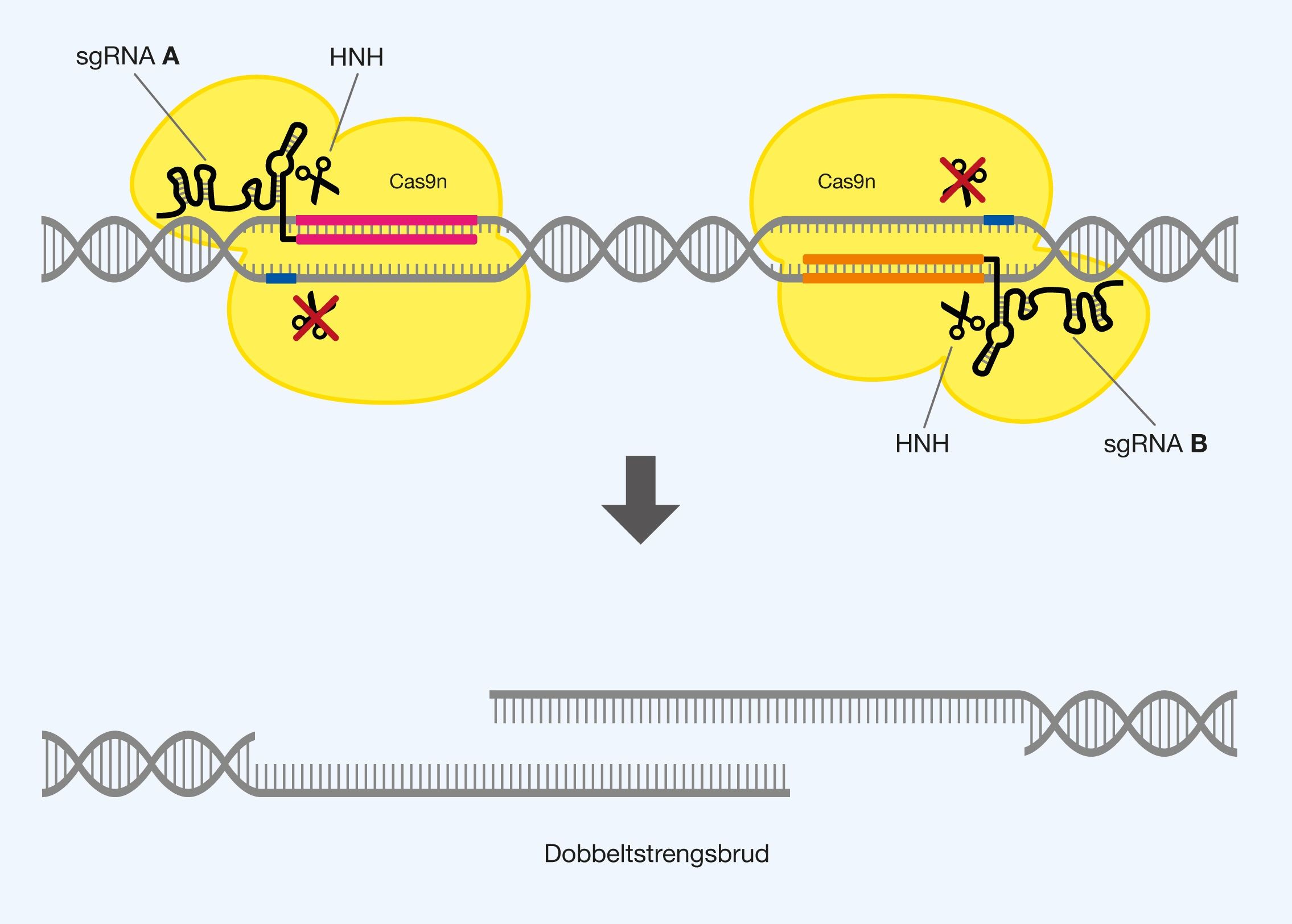
Off-target effekter og mulige løsninger
Det første problem man møder ved genmodifikation med Cas9 er off-target effekter. Disse ses når Cas9-proteinet interagerer med en anden sekvens, end den man havde planlagt at modificere. Det er klart, at dette kan skabe store problemer, da eksempelvis vigtige gener kan blive ødelagt ubevidst. Problemet opstår hovedsageligt fordi sekvensen, som det givne sgRNA genkender, findes flere steder i genomet, eller at der findes sekvenser, der minder tilstrækkeligt om den. De alternative PAM-sekvenser 5’-NAG-’3 og 5’-NGA-3’ kan også blive genkendt i mindre grad, hvilket sænker specificiteten. Off-target effekter er vanskelige at forudse for de enkelte sekvenser i et genom, hvilket gør usikkerheden større ved brugen af Cas9. Man kan reducere mængden af off-target effekter ved at prøve at sikre sig, at sit sgRNA er mest muligt unikt i forhold til én sekvens i genomet. Med specificiteten fra PAM-sekvensen og de 20 genkendende nukleotider, er det let at opnå i organismer med små genomer, men kan være langt mere kompliceret i komplekse organismer med store genomer, såsom mennesker.