RNA Isolering
Denne artikel vil gennemgå RNA-isolering, der er et altafgørende trin, for kvaliteten af en mikrochipanalyse.
Hvorfor isoleres RNA og ikke DNA?
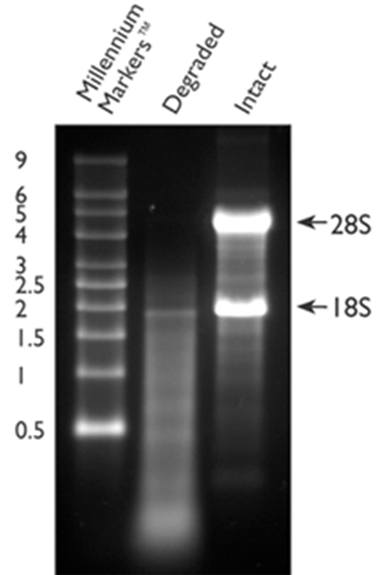
Den store forskel mellem fx en kræftcelle og en almindelig celle ligger ikke i genernes DNA, men derimod i hvilke gener, der transskriberes eller ikke transskriberes – og i hvor ofte de transskriberes. Ved at analysere de mRNA-molekyler, en celle indeholder, kan det afgøres, hvilke gener der bliver transskriberet, og i hvor stort omfang det sker. Når man skal skaffe sig mRNA, vil man oftest benytte celler fra blodet eller celler fra et specifikt væv, hvorfra man udtrækker mRNA vha. specielle teknikker. For at få et vellykket mikrochipeksperiment er det meget vigtigt, at kvaliteten af RNA er god. Med god kvalitet menes, at RNA’et ikke er nedbrudt under isoleringen. Nedbrudt RNA vil i forsøgsresultaterne give et meget højt baggrundssignal og lave værdier af intakt RNA, hvilket helt kan ødelægge den efterfølgende dataanalyse.
Hvordan isoleres RNA?
Kroppens RNA findes inde i cellen. For at få isoleret RNA til eksperimenter skal cellevæggene og plasmamembranen ødelægges, så RNA’et kan komme ud. Her bruger man ofte både en enzymatisk og mekanisk fremgangsmåde. Enzymerne går ind og nedbryder proteinstrukturerne i cellerne ved at nedbryde forskellige bindinger i cellens strukturelle proteiner. Den mekaniske fremgangsmåde kan være sonikering (ødelæggelse vha. lyd), centrifugering, homogenisering med en dounce (kan sammenlignes med en morter) eller ved brug af kanyler. I takt med at cellen ødelægges, frigives ribonucleaser (RNaser), som er enzymer, der nedbryder RNA. Hvis man arbejder hurtigt og samtidigt med den enzymatiske og mekaniske metode, sikrer man sig, at cellevæggene bliver nedbrudt, så enzymet kan komme ind og virke, og man mindsker derved RNA-nedbrydningen.
Ud over cellens frigivelse af RNase, frigiver vi naturligt RNase fra olien på vores hænder, ansigt, hår osv. Desuden kan overflader og støvpartikler indeholde bakterier og virus, som også har RNaser. Det er derfor vigtig, at man arbejder sterilt med specielle RNase-fri handsker, -eppendorf-rør og -pipette-spidser. Holdes prøverne på is vil man med stor sandsynlighed inaktivere RNasen, da enzymet er inaktivt ved dinne temperatur. Der findes også stoffer, som kan inaktivere RNAser, men de er ikke nødvendige, hvis man blot sørger for et sterilt RNase-frit miljø.