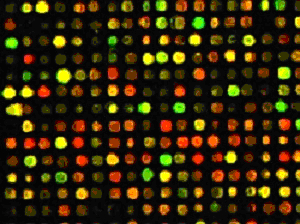
Mikrochipteknologi
Denne artikel omhandler DNA-mikrochipteknologien. Artiklen beskriver, hvordan DNA-mikrochipplatformen er opbygget og hvordan den fungerer. Desuden behandles fluorescens-mærkningen af biologiske materialer.
Hvad kan man bruge en DNA-mikrochip til?
I første halvdel af 1990’erne kunne forskerne kun studere ét gen eller højst en håndfuld af gener ad gangen. Isolering, identifikation og karakterisering af et gen kunne med datidens biologiske værktøjer være ret tidskrævende. Derudover kan det ved analyse af ét gen ad gangen være svært at undersøge samspillet mellem generne, og hvordan dette samspil har betydning i sygdomme.
Det er i dag muligt at undersøge aktiviteten af alle menneskets gener ved brug af DNA-mikrochipteknologi. Det er en teknik, der oprindeligt var udviklet til computerindustrien – deraf ordet chip. I dag bruges mikrochipteknologien primært i forskningen og i medicinalindustrien. Her bruges den til at undersøge funktionen af et gen, dets placering i de biologiske signalveje og til at studere vekselvirkningen mellem flere gener. Derudover kan mikrochipteknologien bruges til at klassificere tumorer baseret på deres genaktivitet, udvikle ny medicin og undersøge toksiske parametre.
Mikrochipteknologien bruges med stor fordel i flere sammenhænge inden for sygdomsforskning. Det er en såkaldt high-throughput screeningsmetode (HTS), da man vha. af en mikrochip kan undersøge tusindvis af faktorer på en gang. Med denne teknologi er det blevet hurtigere og nemmere at få viden om de molekylære ændringer, der kan opstå i menneskets celler som følge af sygdomme, som er relaterede til generne. Dermed er der åbnet for muligheden for at opdage forstadierne til en specifik sygdom og endda måle meget små ændringer i kroppen, inden eventuelle symptomer skulle vise sig.
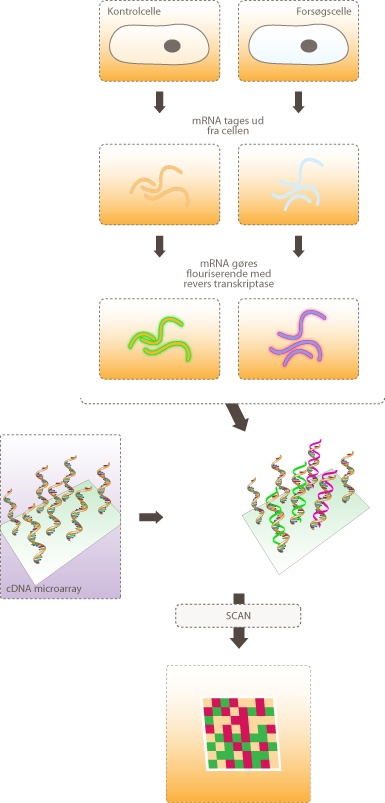
Som en del af denne high-throughput metode måles oftest på flere parametre, flere processer og/eller flere gener for at undersøge en række sammenhænge mellem disse. Et eksempel på, hvordan en mikrochip kan bruges i biologisk sammenhæng, kunne være at undersøge hvilke gener, der påvirkes, når en person har kræft. Dette kan gøres ved at sammenligne en kræftcelle med en almindelig celle fra det samme væv i den samme person. I en sådan model kan man få mere viden om de underliggende mekanismer, der får en normal celle til at forandre sig til en kræftcelle. Man kan tilføje yderligere en parameter ved at tage en vævsprøve fra kræftpatienten før behandling og efter behandling for at undersøge, om aktiviteten af de forskellige gener i kræftcellen har generhvervet de normale aktivitetsniveauer.
En stor forskel ved denne high-throughput metode i forhold til den mere traditionelle ”low-throughput” metode, hvor ét gen undersøges ad gangen, er det enorme mængde datasæt, man får som output. At håndtere et datasæt med flere tusind værdier kræver en systematisk organisering.
For at kunne analysere resultatet fra et mikrochipforsøg kræves, ud over en biologisk viden, også en matematisk viden. Ved at anvende værktøjer, som kan måle gen-information fra det enkelte menneskes celler og kombinere dette med biologisk og matematisk viden, er man i dag blevet bedre til at designe nye og mere avancerede behandlinger mod mange sygdomme. Dette vil ultimativt også åbne muligheden for, at man i fremtiden vil kunne give patienter individualiseret behandling på baggrund af deres personlige genetiske profil. Dermed vil patienterne opnå større overlevelsemulighed og evt. få færre medicinske bivirkninger.
Design af eksperiment
Hvordan skal man designe et eksperiment, når det nu er muligt at måle på aktiviteten af alle menneskets gener? Hvis man leder i blinde blandt 25.000 gener, kunne man bruge meget unødvendig tid på dette uden at blive klogere. Man er nødt til at konkretisere, hvilke spørgsmål man ønsker svar på. Typisk vil man opstille en hypotese, der skal bevises om den er falsk.
Et eksempel kunne være, at man ønsker at få viden om de genændringer, der forekommer i en kræftcelle i forhold til en normal celle. Hypotesen kunne være, at der er forskel på gen-aktiviteten i en kræftcelle i forhold til en normal celle. Her vil man forvente, at de fleste gener ikke viser en forskel i gen-aktivitet, men der vil dog være få gener, som fx proto-onkogener og tumor supressor-gener, som man vil forvente viser forskel i gen-aktivitet.