Signalpeptider og propeptider


Når proteiner er blevet transkriberet og translateret inde i cellen, kan de have mange forskellige destinationer i eller uden for cellen. For at guide dem hen til det rigtige sted, har de fleste proteiner et signalpeptid siddende på sig. Dette signalpeptid medfører, at AMP’er secerneres, dvs. udskilles fra cellen, hvilket er vigtigt, da de udøver deres effekt uden for cellen. Derudover består AMP’er også af en propeptid del der, ligesom signalpeptidet, klippes af, før det sendes ud af cellen. Før propeptidet klippes af, er AMP’et ikke aktivt.
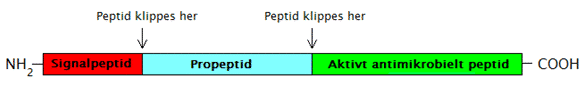
Figur 35. Figuren viser opbygningen af et protein med signalpeptid og propeptid, der begge klippes af for at danne det aktive protein.
AMP’et aktiveres derved først, når propeptidet klippes af, hvilket er smart, da AMP’er er potentielt selvskadelige. De skal derved kun være aktive uden for cellen, og når der er brug for dem, dvs. når der er bakterier til stede.
Signalpeptidet, der medfører, at AMP’er secerneres findes altid i starten (den N-terminale ende) af peptidet (figur 35).
Signalpeptidet opfattes af cellen ved, at et specifikt signal-genkendelsesmolekyle (signal recognition particle (SRP)) bindes til signalpeptidet på det nye protein. Når SRP er bundet til signalpeptidet, vil translationen af mRNA til protein midlertidigt stoppe. SRP og det ufærdige protein vil dernæst bevæge sig hen til det ru endoplasmatiske retikulum (ruER) (figur 36), hvor SRP binder til en SRP-receptor på det ruER. Grunden til, at det kaldes det ru ER er, at der sidder ribosomerpå det, hvorved overfladen ser ujævn ud. Derudover sidder der ribosombindende receptorer på det ruER, hvilke ikke er til stede på det glatte ER. Ribosomreceptorerne binder ribosomerne, hvortil der er bundet det ufærdige protein og signalpeptid. Når SRP er bundet til en SRP-receptor på det ruER samtidig med, at ribosomet er bundet til en ribosomreceptor, kan translationen genoptages. Translationen færdiggøres, hvorefter ribosomet vil falde af det ruER. Ribosomet kan nu binde et nyt stykke mRNA, der skal translateres.
Figur 36. Det ru endoplasmatiske retikulum (ruER), hvortil ribosomer er bundet, indgår i syntesen af proteiner, modificering af proteiner og dannelsen af cellemembranen.
Idet SRP binder til en SRP-receptor på det ruER, vil SRP frigøres, hvorefter translationen kan fortsætte. Det nye peptid syntetiseres direkte ind i det ruER. Inde i ER findes enzymet, signalpeptidase, der klipper signalpeptidet af proteinet. Dét sted, hvor signalpeptidasen klipper signalpeptidet fra, kaldes for kløvningssitet. Når signalpeptidet er blevet klippet fra, er peptidet klar til videre distribution i cellen eller til videre transport ud af cellen via golgiapparatet (figur 37). Peptidet vil blive transporteret af transportvesikler fra det ruER til cis siden af golgiapparatet. I golgiapparatet kan der evt. ske videre modifikation af peptidet. Dernæst vil peptidet blive transporteret i vesikler ud til cellemembranen, hvor de via exocytosebliver secerneret ud af cellen.
Som tidligere beskrevet, er det vigtigt at AMP’er har en signalsekvens, der dirigerer dem ud af cellen. Dette skyldes, at AMP’er udøver deres effekt uden for cellen, hvor de skal slå mikrober ihjel. Center for Biologisk Sekvens Analyse (CBS) ved Institut for Systembiologi på DTU benytter sig af bioinformatik og har udviklet et program, SignalP, som kan forudsige signalpeptider og deres kløvningssites.
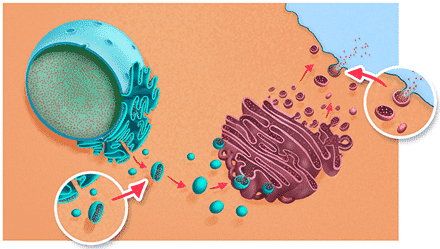
Figur 37. Figuren viser en del af en celle og nogle af organellerne, der findes i cytoplasma. Golgiapparatet ses på højre side af figuren. Dets primære funktion er at færdiggøre proteiner, så de er klar til eksport ud af cellen.
Inden for bioinformatikken findes der mange forskellige værktøjer, som kan benyttes hvis man er på udkig efter nye AMP’er. En strategi kunne være at lede efter peptider, hvortil signalpeptider er bundet, og derefter se om disse peptider er AMP’er. Man gør herved sin søgning efter AMP’er meget simplere, idet man først identificerer proteiner med signalpeptider, før man begynder at lede efter AMP’er blandt disse. Tænk på, at der er mange tusinde gener i eukaryoter, så hvis man vil finde nye AMP’er til medicinsk forskning, er det noget lettere, hvis man kun leder efter AMP’er blandt de gener, der secerneres. Proteiner, der secerneres, er kun en brøkdel af det samlede antal proteiner i cellen. Man kan herefter bruge SignalP til at verificere tilstedeværelsen af et signalpeptid og afgøre, hvor kløvningssitet for signalpeptidet sidder. Det ses herved, at man ved simpel hjælp fra bioinformatikken kan reducere sin AMP-eftersøgning ganske dramatisk i de ellers uoverskuelige store mængder af DNA sekvenser.
For at demonstrere hvordan bioinformatik kan tages i brug, kan du selv prøve at benytte SignalP til at forudsige positionen af kløvningssitet for et signalpeptid siddende på et AMP. Lad os vælge at undersøge Plectasin, som blev opdaget af Novozymes i 2002 og viste sig at være et meget lovende antibiotikum, der var effektivt mod multiresistente bakterier.
Gå først til SignalP ved at klikke her.
Nedenfor ses aminosyresekvensen for Plectasin. Kopier denne og sæt den ind i det tomme felt i SignalP.
Klik herefter ’Submit’ og læs resultaterne igennem. Hvad står der om signalsekvensen, og efter hvilken aminosyre i sekvensen findes kløvningssitet? Hvad står der efter ”Cleavage site between…”?
Vi kan undersøge om programmet har fundet det rigtige kløvningssite ved at benytte databasen UniProt. I søgefeltet skrives ’Plectasin’, hvorved der fås et enkelt hit, som hedder Q53I06 (PLECT_PSENR). Klik på dette hit og kig under Sequence annotation (Features), hvor du finder en liste over længden (antal aminosyrer i sekvensen) af signalpeptidet, propeptidet og den aktive peptidkæde. Her kan du se, om længden af signalpeptidet stemmer overens med dét, du fandt ved at benytte SignalP.