Begrebet metabolic engineering dækker over en række moderne analytiske og praktiske metoder inden for den anvendte molekylærbiologi, hvormed man modificerer de reaktionsveje, som en mikrobiel celle benytter. Det kan f.eks. glukolysen, citronsyre-cyklen eller andre af cellens stofskifteprocesser. Modifikationen sker ved en indsættelse eller fjernelse af gener, og hensigten er at forbedre cellens egenskaber, f.eks. i forhold til at øge produktionen et givent stof.
Formålet med en sådan modificering er lettest af forstå ved et eksempel. I et tidligere afsnit nævntes det, at gær ikke kan nedbryde og derved anvende pentoser. Et sådant problem ville man tidligere ikke have haft chance for at løse. Men med matematikkens og computerens indtog i biologiens verden er vi trådt ind i den såkaldte post-genome æra. Vi har nu kendte genomsekvenser for et stort antal forskellige organismer, og det har gjort det muligt, at finde frem til de gener som skal til, for at en given organisme kan optage og bruge xylose. Ved hjælp af metabolic engineering kan disse gener indsættes i gærs genom, og den resulterende stamme undersøges mht., om den kan bruge xylose.

Figur 13. Scanning elektronmikrograf af celler af Saccharomyces cerevisiae (Wheals 2002).
Metabolic engineering er det overordnede begreb – det integrerer analysen af cellen, den matematiske behandling af data og det efterfølgende eksperimentelle forsøg for at karakterisere den opnåede fænotype og evaluere resultatet i virkelighedens verden. De opnåede erfaringer kan så igen indgå til finjustering af modellen, hvorved der effektivt opstår en såkaldt iterativ cyklus til gradvis forbedring mod målet (se figur 14). Industriel bioteknologi benytter metabolic engineering som vejen til at udvikle grønne cellefabrikker.
LÆS MERE
Eksperter i cellefabrikker
Danske Fluxome Sciences er eksperter inden for industriel bioteknologi og udvikler innovative og kosteffektive bioprocesser vha. metabolic engineering. Firmaet har blandt andet foretaget dybdegående analyser af ethanoldannelsen i gær – i en såkaldt genome scale model – og har derved kunnet forbedre udbyttet med 5 % (hvilket er meget for denne proces).
Kæmpe databaser der kan bruges til metabolic engineering
Store databaser med biologisk information eksisterer i dag i takt med at flere og flere organismer sekventeres (deres genomers sekvens bestemmes). Prøv at søge efter ’xylose’ i KEGG eller efter ’TEF1’ i Saccharomyces Genome Database
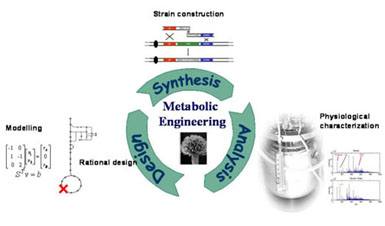
Figur 14. Et billede af den såkaldte iterative cyklus indenfor metabolic engineering. Der er tre delmomenter. Først designes stofskiftet via en computermodel og biokemisk information (Design). Dernæst udføres de genetiske manipulationer, som er blevet forslået i design-fasen (Synthesis = syntese). Endelig analyseres det fremkomne resultat; virkede ændringerne efter hensigten? (Analysis = analyse).

