Bioteknologiske metoder til at undersøge evolution


Fylogeni er en beskrivelse af evolutionshistorien og slægtsforholdene mellem forskellige organismer. Et fylogenetisk træ illustrerer disse forhold i et diagram, hvor hvert forgreningspunkt i træet angiver en teoretisk fælles ”forfader” for enhederne længere ude af grenen.
Protein- eller DNA/RNA-sekvenser er særdeles velegnede til at opstille de evolutionære forhold, idet man ved sammenligning af sådanne sekvenser kan se, hvor nært beslægtede organismer er. Jo længere tid organismerne har udviklet sig hver for sig, jo mere forskellige er de. I figur 11 ses et eksempel på et fylogenetisk træ.
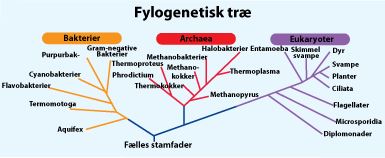
Figur 11. Skitse af et fylogenetisk træ.
Inden man kan sammenligne gener eller proteiner mellem forskellige organismer, er det nødvendigt at kunne identificere dele af molekylerne som oprindeligt har været ens. Dette stiller krav til det gen eller protein der anvendes. Med udgangspunkt i genet skal det først og fremmest være til stede og have samme funktion, i alle organismer der undersøges. Det skal sikre at dele af genet er konserveret dvs. uændret på tværs af de forskellige organismer. Derudover er det også nødvendigt, at der er dele af genet som er stærkt varieret mellem forskellige organismer, således at der kan kendes forskel på to nært beslægtede arter. Kravene til anvendelse af gener i evolutionære studier er opsummeret i det følgende:
Krav til gener for anvendelse i evolutionære studier
Gensekvensen for Ribosomalt RNA (rRNA) er den hyppigst anvendte DNA-sekvens til konstruktion af fylogenetiske træer, da genet opfylder de beskrevne krav (se ovenfor).
Ribosomalt RNA, rRNA
rRNA er hovedelementet i ribosomer og har derfor en vigtig rolle i proteinsyntesen. Alle levende organismer indeholder rRNA. rRNA findes i store mængder i cellerne, da det sikrer at organismerne kan have mange ribosomer og opretholde en hurtig dannelse af proteiner. Ribosomet består af fire rRNA subunits samt en masse proteiner. For prokaryoter analyseres især 16S rRNA (i eukaryoter 18S rRNA), som er RNA-delen af den lille subunit til ribosomet. Dette gen har været anvendt da det har en passende størrelse. Det er relativt hurtig at sekventere og langt nok (ca. 1550 nukleotider) til at give en pålidelig fylogenetisk analyse.
Strukturen af et 16S ribosomalt RNA er meget specifik og dannes ved hjælp af baseparring, inverted repeats og loops. Man kan sammenligne forskellige sekvenser af 16S rRNA ved hjælp af bioinformatik-programmer. I eukaryoter findes en lignende subunit, som kan sekventeres og analyseres. Denne er dog lidt større og betegnes 18S rRNA.
Før man kan sammenligne sekvenser af 16S rRNA fra forskellige organismer, er det naturligvis nødvendigt at sekventere genet. Inden sekventering er det nødvendigt at opformere genet, som gøres ved hjælp af en teknik der hedder PCR (beskrives senere).
Ved at sammenligne 16S rRNA sekvenser kan man adskille organismer på artsniveau og opefter. Det er således ikke muligt at se forskel på to individer inden for samme art ved at analysere de to 16S rRNA sekvenser.
Før man benyttede fylogeni, som dannes ud fra kendte gensekvenser, var taxonomi en metode, hvormed man kunne klassificere organismer. Taxonomien bygger på sammenhænge mellem organismers karaktertræk, dvs. organismernes fænotype, mens fylogenien bygger på genotype. Der vil i mange tilfælde være sammenhæng mellem taxonomi og fylogeni, men det er ikke altid sikkert at de følges ad. To organismer der i lang tid har været gennem forskellige evolutionsforløb vil være placeret langt fra hinanden i det fylogenetiske træ. De samme to organismer kan godt have fælles karaktertræk og blive placeret tæt på hinanden i taxonomien. For eksempel mente man tidligere at svovlbakterier og cyanobakterier var nært beslægtede da de begge har fotosyntese. Efter at have sekventeret deres 16S rRNA og opstillet dem i et fylogenetisk træ, er det imidlertid vist, at de to bakterier befinder sig meget langt fra hinanden i evolutionsforløbet.
PCR er en forkortelse af det engelske navn “Polymerase chain reaction”, og som navnet indikerer, er der tale om en kædereaktion.
PCR er en metode der blandt andet bruges til at opformere dele af DNA-sekvenser in vitro (latin for “i glas”. Dvs. ikke i kroppen). Ved hjælp af specifikke primere og en termostabil DNA-polymerase dannes store mængder af specifikke gener.
PCR består af mange cykler hvor en række trin gentages. Hvert trin har en specifik temperatur i et vist tidsrum. Hver cyklus svarer til en DNA-replikation, hvilket vil sige at mængden af DNA fordobles.
Første trin i PCR-maskinen består i opvarmning af DNA. Opvarmningen ødelægger hydrogenbindinger og van der Waals kræfter, hvilket forårsager at DNA denaturerer. Ved denaturering forstås at DNA mister sin sekundære struktur og bliver til to enkeltstrenge i stedet for en dobbeltstrenget α-helix.
Derefter binder to primere sig til hver sin DNA-streng efter baseparringsprincippet. En primer er en kort DNA-streng, som er komplementær til DNA-skabelonen. Denne del er nødvendig for at DNA kan replikeres da DNA-polymerasen skal bruge et stykke DNA at starte på og ikke en nøgen streng. Primerne er tilsat PCR-blandingen inden start.
Primerne binder ved at PCR-blandingen nedkøles. Hydrogenbindinger og van der Waals-kræfter bliver aktive igen. Der er tilsat et stort overskud af primere i blandingen for at sikre at DNA-strengene binder til primere og ikke til hinanden. DNA-polymerasen syntetiserer DNA når blandingen varmes en anelse mere op igen. DNA-polymerasen benytter DNA-strengene som skabelon og syntetiserer den komplementære streng. Nukleotiderne dATP, dTTP, dGTP og dCTP fungerer som byggesten og skal derfor også være til stede i PCR-blandingen. Efter én cyklus er antallet af den specifikke DNA-sekvens fordoblet. Nu kan proceduren gentages gang på gang. Normalt udføres 30-40 cykler i en PCR-maskine. Ved 30 cykler fordobles DNA’et 30 gange, og der opstår derved 230 kopier af det oprindelige stykke DNA.
Opvarmningen i PCR-maskinen er nødvendig for at denaturere DNA. Det er derfor vigtigt at DNA-polymerasen ikke ødelægges ved høje temperaturer. Det er løst ved at isolere DNA polymerasen fra bakterien Thermus aquaticus der lever under varme forhold. Denne polymerase kaldes Taq polymerase og er stabil op til 95 °C.
Dette er et klassisk eksempel på hvordan forskere i områder med ekstreme forhold, har fundet enzymer med ønskede egenskaber.
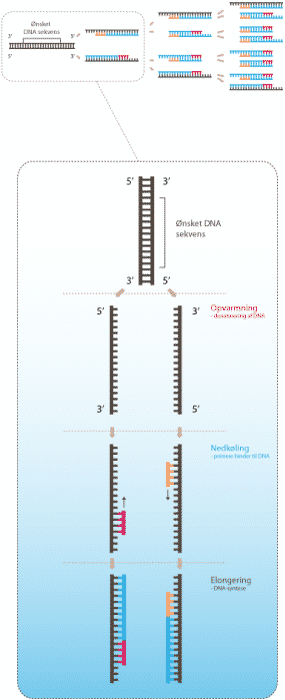
Figur 12. Illustration af PCR. Den øverste del af figuren viser de første 3 cykler i en PCR-reaktion. Den nederste del viser detaljerne bag en cyklus. Først denatureres DNA ved opvarmning. Herefter følger en nedkøling hvor primere binder til hver sin DNA-streng. Derefter kopiers DNA-strengene.
Når man laver en sekventering, ønsker man at finde rækkefølgen af baserne i et stykke ukendt DNA. En DNA-sekvens er netop rækkefølgen af baserne for et bestemt stykke DNA.
Flere metoder til sekventering af DNA findes. Metoder adskiller sig ved hvor lange stykker af DNA de kan aflæse af gangen, samt hvor mange baser der læses per tidsenhed. En af de første, og meget anvendte metoder til sekventering, er Sanger sekventering.
Ved Sanger sekventering benyttes også en PCR-reaktion, og princippet er det samme som ved opformering af DNA. Der tilsættes DNA-polymerase, primer (kun én primer) og nukleotider (dNTP, hvor N står for en nukleobase, dvs. enten A, T, C eller G) samt nogle modificerede nukleotider, som stopper replikationen. Disse stop-nukleotider er di-deoxynukleotider (ddNTP) som er mærket med hver sit fluorescerende stof der kan detekteres. Når PCR-reaktionen er løbet til ende, vil der pga. stopnukleotiderne være opstået DNA-fragmenter med forskellig størrelse. Alle fragmenterne starter det samme sted, nemlig med den primer man har anvendt i reaktionen. Fragmenterne stopper med et ddNTP der fluorescerer i en farve. Farven angiver hvilket nukleotid der er på den pågældende position. DNA-fragmenterne kan adskilles efter størrelse. Enten ved hjælp af en elektroforese-gel eller ved kapillar elektroforese som er det mest benyttede i dag. Det bagvedliggende princip er det samme. Fragmenterne vil trænge igennem gelen med forskellig hastighed – de små hurtigst og de store langsomst. Nu kan sekvensen af DNA aflæses, ved at se hvilken farve de enkelte bånd fluorescerer. Det afslører hvilket stopnukleotid der er tale om. I figur 13 vil det DNA-fragment der trænger hurtigst gennem gelen være mærket med en blå fluorescens, som svarer til basen A, og det kan derfor konkluderes at det første bogstav i sekvensen er A.

Figur 13. Princippet bag Sanger sekventering. Fire PCR reaktioner opsættes, én for hver mulig stop-nukleotid (ddNTP). Stop-nukleotiderne inkorporeres tilfældigt i den voksende DNA-kæde, hvilket terminerer replikationen.
Hvert stop-nukleotid kan fluorescere med en specifik farve, der kan aflæses af en maskine. Dette giver et datasæt som vist i figur 14.
Det er ofte robotter der udfører DNA-sekventering. Detektionen fungerer ved at der sendes laserstråler mod DNA-fragmenterne, hvilket giver et bestemt udslag af fluorescens. Signalet af fluorescens hørende til et bestemt DNA-fragments stopnukleotid opsamles i et datasæt på en computer.
Et eksempel på et udsnit af sådan datasæt ses i figur 4. De forskellige toppe viser hvilket nukleotid der er blevet detekteret i den givne position. Nogle steder kan der opstå tvivl om hvilken base der er korrekt. Det ses ved at der fremkommer overlappende toppe. Grunden til at der fremkommer to toppe oven i hinanden er at teknikken ikke er perfekt. Det kan f.eks. være svært at adskille meget små fragmenter fra hinanden, og de kan derved blive detekteret samtidigt.
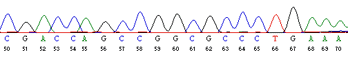
Figur 14. Eksempel på datasæt for Sanger sekventering. Hver base giver et bestemt flourescens-udslag der aflæses. Dette afslører sekvensen af nukleotider i DNA’et.
Højden af toppene indikerer hvor stærkt detektionssignalet er. Når der kun er en top til stede, vil det ikke spille nogen rolle. Hvis der derimod fremkommer to toppe, hvor den ene er lav, og den anden er høj, vil computeren indsætte basen for den højeste top på denne position. Computeren vil i dette tilfælde markere positionen med en gul streg, så det er muligt at se at der er to muligheder for den givne position.
Andre metoder til sekventering bygger på nyere teknologier, der tillader sekventering af hele genomer på ganske kort tid. Eksempelvis Illumina sekventering, SOLiD sekventering og Nanopore sekventering.
Når DNA er sekventeret, er det muligt at sammenligne med andre DNA-sekvenser ved at sammenholde sekvenserne base for base. Denne sekvenssammenligning kaldes på engelsk for sequence alignment.
Hvis sekvenserne ikke er lige lange, eller når der er brug for det, indsættes gaps som forlænger et stykke af sekvensen. Hvis de ikke blev indsat, ville to relativt ens molekyler se ud til at være vidt forskellige. Indsættelsen af gaps er illustreret i figur 15. I figur 16 er vist hvordan et sequence alignment output i praksis kan se ud.
Når man har at gøre med små DNA-stykker, vil man godt kunne udføre denne sammenligning ved selv at kigge på sekvenserne, men som regel er DNA-sekvenserne så lange, at det er nødvendigt at bruge en computer. For eksempel er DNA-sekvensen for 16S rRNA ca. 1550 nukleotider lang.
Der er udviklet computerprogrammer til at lave disse sekvenssammenligninger, og de kan godt sammenligne mange sekvenser på en gang. Ud over at indsætte gaps er programmerne lavet til at genkende de vigtigste områder på sekvenserne. Disse dele kaldes ankerpositioner. Når sekvenssammenligningerne er gennemført, er det muligt at se hvor meget sekvenserne hver især ligner hinanden, og derudfra opstille fylogenetiske træer. Hvis to sekvenser er meget ens, skal de ligge tæt ved hinanden på træet.

Figur 15. Sequence alignment med indsættelse af gaps. Et-tallerne viser når der er overensstemmelse mellem de to sekvenser, mens 0’erne viser at der ikke er overensstemmelse. Det ses at efter indsættelse af gaps viser de to sekvenser ret stor lighed.

Figur 16. Sequence alignment output. Stregerne angiver hvor der er sammenhæng mellem de to sekvenser.