Sekventering
Sekventering henviser til en række af forskellige teknologier, der relativt hurtigt kan kortlægge rækkefølgen af baserne (A, C, G, T/U) i genomet hos en bestemt organisme. Der findes forskellige typer og generationer af sekventeringsteknologier, der kendetegnes ved, hvad man ønsker at undersøge. Man kan groft opdele teknologierne i to kategorier – hvis man ønsker at sekventere en organisme der allerede er kendt eller en der er ukendt. Hvis man ønsker at undersøge en organisme der allerede er sekventeret før, er det ofte for at undersøge for mutationer eller variationer (ondartet eller godartet ændring i DNA’et). Dette kunne f.eks. være, når en person er syg og man ønsker at finde de DNA-ændringer der har medført sygdommen, eller til at undersøge om forskellige personer er i familie sammen. Hvis man ønsker at sekventere en hel ny organisme (DNA-sekvenser man ikke har set før), er det for at finde nye gener og stoffer. F.eks. hvis en bakterie er modstandsdygtig overfor en bestemt type antibiotika og man ønsker at finde resistensgenet. Eller som ved ARGO hvor de sekventerer planten Jaborandi for at finde de gener der danner stoffet pilocarpin, der kan bruges til at behandle grøn stær. Man kan altså bruge sekventering i alverdens sammenhænge – lige fra at definere evolutionære forhold, til at finde sekundære metabolitter som kan bruges til behandling af sygdomme.
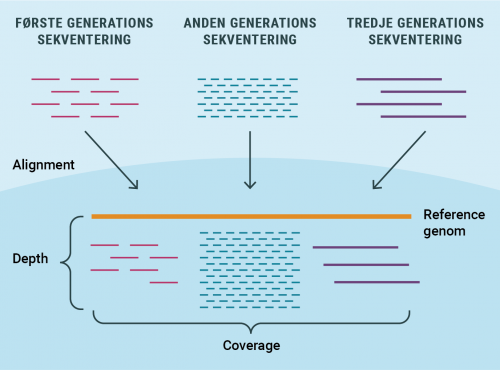
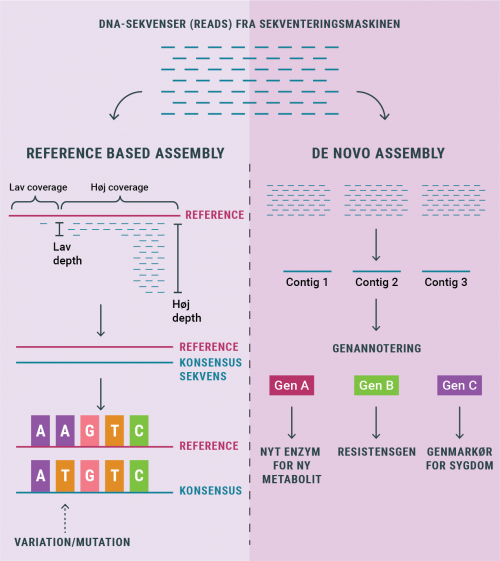
Der findes tre generationer af sekventeringteknologier: første-, anden- og tredje generations sekventering, de bruges på forskellige områder med hver deres fordele og ulemper. Fælles for dem alle er at de producerer DNA-sekvenser, også kaldet reads, som efterfølgende skal sættes sammen ved assembly. En forståelse for hver generation, samt hvor og hvornår de benyttes, er essentiel når man skal afgøre hvilken slags data man bygger sine analyser på.
Første generation sekventering – Sanger sekventering
Den første generation af sekventering kaldes Sanger sekventering, chain-termination metoden, eller dideoxynukleotid metoden. Teknikken er langsom og laboratorieintensiv, men producerer reads med en længde mellem 600 og 1000 basepar (bp) med en forholdsvis lav fejlrate. Selvom teknologien er blevet erstattet af nyere generationer, benyttes metoden stadig ved kortlægning af enkelte gener, f.eks. hvis man genmodificerer en plante og vil teste at genet er indsat og placeret korrekt. Sanger sekventering bruges også ved et mindre antal af prøver eller til at validere reads produceret fra senere generationer af sekventering.
Anden generation sekventering – Next-Generation Sequencing
Next-Generation Sequencing (NGS) er nutidens mest udbredte metode og markedet er hovedsageligt domineret af firmaet Illumina. Metoden producerer rigtig mange reads, virkelig hurtigt og billigt. Desværre er DNA fragmenterne meget korte, hvilket gør det til en endnu større udfordring, når disse skal assembles til et fuldt genom. Et assembly kan sammenlignes med at samle et kæmpestort pulsespil, hvor nogen brikker mangler og andre kan være ens, og i tilfælde af at det er en ny organisme (de novo), ved man heller ikke hvordan puslespillet skal se ud. NGS bruges i alle tilfælde, hvor man har mange prøver eller store genomer. F.eks. hvis man sekventerer hele genomet fra en syg person, for efterfølgende at kunne analysere hvad personen fejler.
Tredje generation sekventering – PacBio / Nanopore
Ved tidligere generationer af sekventering skal DNA først brydes i fragmenter og derefter amplificeres ved PCR for at kunne blive sekventeret. Dette er ikke nødvendigt ved tredje generations sekventering (TGS), der kan kortlægge et helt enkelt molekyle af DNA. Dette har medført to vigtige fordele: der produceres meget lange reads og baserne bestemmes med det samme, når de forløber i sekventeringen (realtime). Dette er modsat ved Sanger og NGS, hvor man er nødt til at vente til reaktionerne er kørt færdig for at se rækkefølgen af baserne. TGS-teknologierne er først blevet præsenteret på markedet i løbet af dette årti, og der er to metoder der på nuværende tidspunkt dominerer området: Pacific Biosciences (PacBio) der bruger metoden Single-molecule real-time (SMRT) og Oxford Nanopore Technologies som bruger Nanopore Sequencer. TGS er på mange måder fordelagtigt over NGS og Sanger, men der produceres ikke lige så meget data som ved NGS, og der fås en højere sekventeringsfejlrate. Derfor kombinerer forskere ofte forskellige generationer af sekventering. TGS kan give lange reads, som kan hjælpe med at dække områder af genomet med høje repetitive regioner, mens NGS kan give en høj dækning i dybden (uddybes senere).