FigTree er et program der benyttes til at visualisere fylogenetiske træer, så man blandt andet kan danne sig et overblik over organismers slægtskab (find det her). FigTree bliver også brugt i undervisningsforløbet: Bioinformatik – En introduktion.
Beskrivelse
FigTree bruges til at visualisere slægtskaber mellem organismer gennem fylogenetiske træer. Fylogenetiske træer (se figur 1) kan konstrueres på forskellige måder. Hver måde benytter en bestemt matematisk model, og valg af denne vil bestemme, hvordan slægtskabet mellem organismerne bliver. Oftest er der ikke variation i det overordnede slægtskab, men det er vigtigt at holde sig for øje, at et evolutionært slægtskab ikke altid er entydigt. I øvelserne er valg af matematisk model underordnet, da FigTree bruges til at visualisere træer, der er lavet på forhånd.
Generel guide
Inden man kan benytte FigTree, skal man have lavet et træ og gemt det i phylip-format. Et træ kan laves med programmerne ClustalX eller Treehugger, men i øvelserne der omhandler FigTree, får du træer, der lavet på forhånd, og du skal derfor ikke selv konstruere dem.
I et åbnet FigTree-vindue benyttes open til at åbne phylip-filen med det træ, man vil visualisere. Træet, der vises, har en rod, hvilket betyder, at den ældste stamfader er valgt. Roden er dog valgt tilfældigt af FigTree og giver derfor ikke et korrekt billede af det kronologiske slægtskab. For at få et rigtigt billede af slægtskabet bør træet vises uden rod. Et ikke-rodet træ viser nemlig, hvordan træets taxa er relateret til hinanden, men giver ingen indikation om kronologien i slægtskabet, dvs. træet giver ikke nogen informationer om, hvilke taxa der er ældst eller yngst. I figur 2 kan det ses, hvordan man viser et træ uden rod.
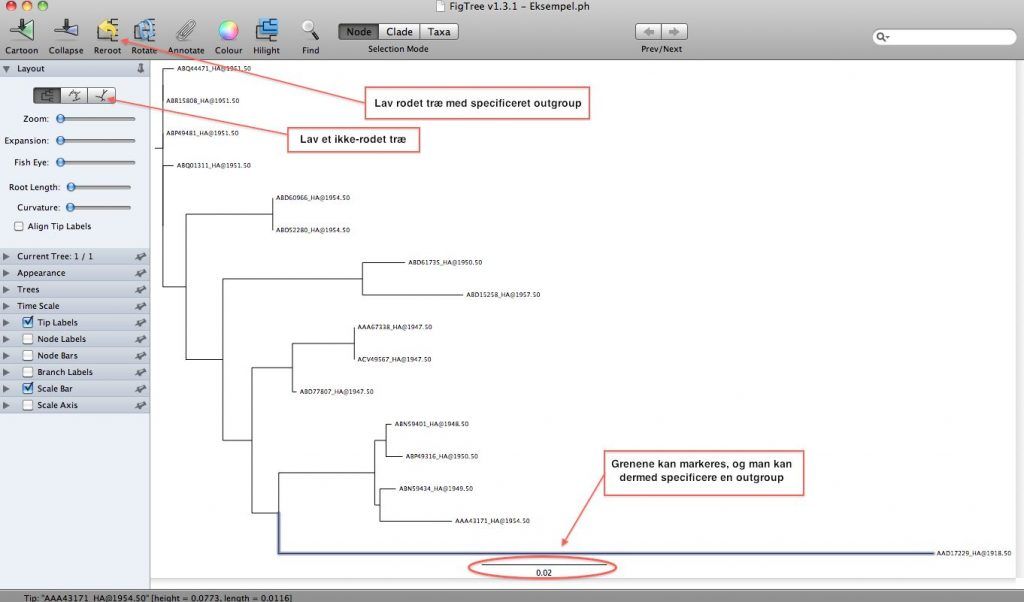
For at lave træet med en korrekt rod skal man have kendskab til de forskellige taxa i træet og vide, hvilken der er fjernest beslægtet. Hvis man ved hvilken taxon, der er fjernest beslægtet med de andre, kan en ydergruppe vælges, og træet vil kunne rodes i forhold til den. Ved at vælge en ydergruppe specificerer man, hvilken taxon der er længst fra de andre, og FigTree vil lave et nyt træ, som placerer ydergruppen længst væk fra de andre taxa.
Træet omrodes ved først at trykke på denne taxon, der skal være ydergruppe og derefter benytte kommandoen Reroot, se figur 2.
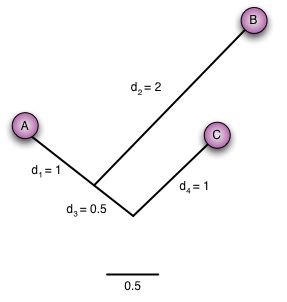
Grenene mellem de forskellige taxa i træet svarer til den evolutionære distance mellem dem, dvs. forskellen målt i f.eks. antal mutationer. Længden er defineret som antal mutationer divideret med længden af sekvenssammenligningen (alignmentet) inklusiv gaps mellem de to taxa. Det skal påpeges, at grenlængden ikke indikerer, hvor tæt beslægtet de forskellige taxa er med hinanden. I figur 3 ses det, at taxon A og B er tættest beslægtet, selvom grenlængden mellem dem (1+2 = 3) er længere end den mellem A og C (1+0.5+1 = 2.5). Grunden til dette er, at sekvensen for A er tættere på skevensen for C end den er på B. Dette kan f.eks. skyldes, at B har akkumuleret flere mutationer efter den divergerede væk fra A i forhold til antal af mutationer, stamfaderen til A og B fik efter C divergerede væk.
Målestokken er også vigtig at kigge på, da den angiver antal mutationer pr. afstand. En høj målestok er således ensbetydende med mange mutationer og dermed stor forskel mellem træets taxa.
Grenlængde og målstok er således vigtige for at kunne fortolke slægtskabet korrekt og ikke drage forhastede konklusioner om et eventuelt tæt slægtskab. I figur 2 er vist, hvor man kan finde målestokken i FigTree. For figur 3 gælder det at: på trods af, at grenlængden mellem A og B er større end mellem A og C, er A og B faktisk tættere beslægtet, dvs. de to taxa divergerede senere væk fra hinanden end C gjorde fra deres stamfader. Grunden til at grenlængden mellem A og C er kortere er, at deres sekvenser er tættere på hinanden end A og B. Dette kan eventuelt skyldes, at B har muteret mere siden den divergerede fra A.